文獻解讀|IF=11.878,前列腺癌生物學和基因調控機制
信息來源:金開瑞 作者:genecreate 發布時間:2019-11-15 16:03:49
題目:A high-resolution 3D epigenomic map reveals insights into the creation of the prostate cancer transcriptome
期刊:nature communications
期刊:nature communications
影響因子:11.878
主要技術:Hi-C、ChIP-seq、RNA-seq、NOMe-seq
研究背景
前列腺癌是主要的新癌癥,是男性的第二大常見癌癥和全球第四大常見腫瘤類型。與正常前列腺細胞不同,前列腺癌細胞轉錄極大失調,細胞增殖不受控制。因此,新發現的控制前列腺癌轉錄組機制將為診斷和治療提供有價值的信息。
人類基因組在細胞核的三維(3D)空間中分為活躍和不活躍的區室,高接觸頻率的區域稱為拓撲關聯域(TAD)。隨著細胞從一種類型變為另一種類型,部分TAD會在活躍和不活躍染色質狀態之間轉換,TAD的狀態改變會影響TAD內基因的表達水平,而且基因通常是由位于同一TAD中的增強子調控的。在由CTCF結合的調控元件對之間的染色質環改變,可能會使遠距離調控元件的啟動子聚集在一起(或分開),從而引起基因表達的變化。因此,轉錄組可能會受到TAD邊界變化的影響。通過原位Hi-C技術,結合ATAC-seq或NOMe-seq(核小體占據和甲基化測序)等方法從中推斷出染色質相互作用中涉及的TF,定義了與TF相對應的核小體耗盡區(NDR)。本文研究了a.TAD的表觀遺傳狀態,b.識別并表征正常和癌癥特異性的增強子-啟動子環。TAD和染色質環的這種特征提供了更詳細的前列腺腫瘤發生的表觀遺傳機制。
研究思路:
研究結果
1、導致轉錄組變化的癌癥特異性TAD
作者在正常前列腺(RWPE1)細胞、前列腺癌(C42B 和 22Rv1)細胞使用原位Hi-C構建了高分辨率的染色質互作數據。在所有三種細胞系中,作者發現3937(?60%)的TAD具有邊界(圖1b)。這些常見的TAD的平均大小約為350 kb(圖1c)。作者發現1328個TAD在RWPE1細胞中檢測到的,但未在任一癌細胞系中檢測到;有1162個TAD在兩個癌細胞系中有但在正常細胞中均未檢測到的。有趣的是,正常細胞的特異性TAD通常大于癌癥細胞的特異性TAD。
TAD的大小變化可能與基因表達變化有關,比較正常細胞和癌細胞全基因組之間的TAD大小,在正常細胞中鑒定出約520個大TAD,相當于癌細胞中約850個較小TAD。與正常細胞相比,在這些改變的TAD中,在癌細胞中表達的基因相對更多(圖1d)。較小的TAD中的許多基因具有更高的轉錄活性,這表明較小的TAD會將基因與抑制元件隔離開來。
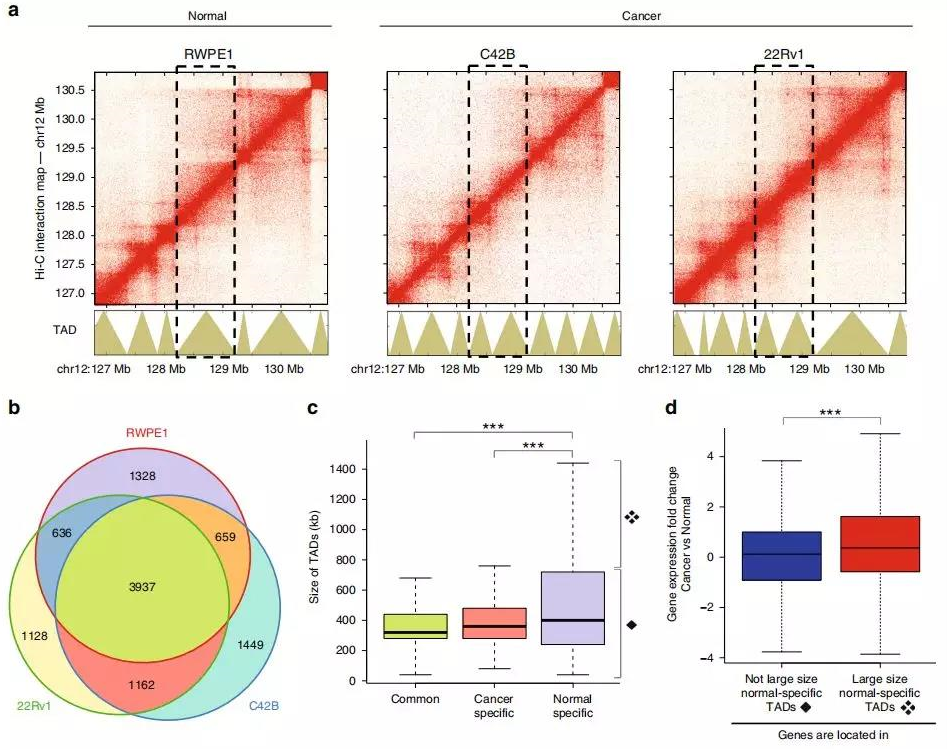
Fig. 1 TAD邊界的改變導致前列腺癌轉錄組的改變
2、可改變染色質狀態的普通TAD
為研究TAD染色質狀態的整體性質和表觀遺傳狀態如何影響TAD內基因表達水平的信息,作者在正常細胞和前列腺癌細胞中使用 ChIP-seq進一步研究。研究發現在正常細胞中的大的TAD可能在腫瘤轉化過程中分裂為較小的活性TAD,從而為以上提出的假說提供了支持,即正常特異性大的TAD可能包含使基因低表達的抑制因子。
此外,作者研究了在正常細胞和癌細胞之間共享邊界的TAD。在變換了狀態的TAD中確定約2000個基因,其中> 40%的基因表達水平顯著改變;與未改變狀態的TAD相比,改變的TAD中差異表達基因的數量增加了約50%。
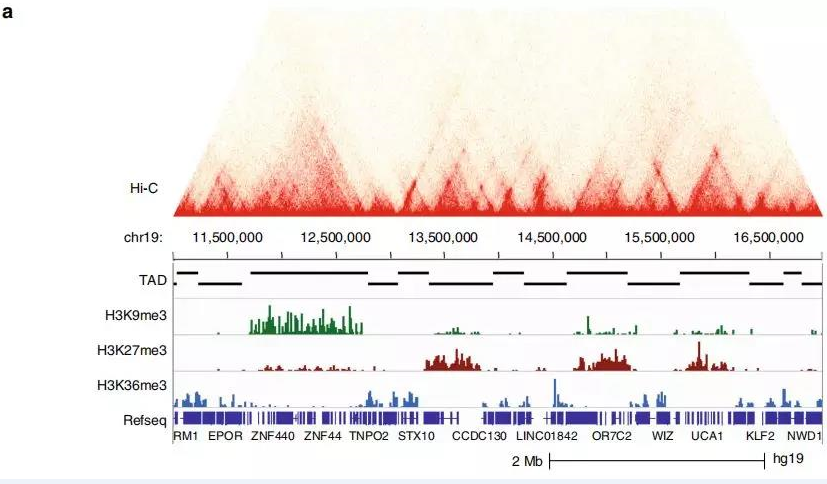
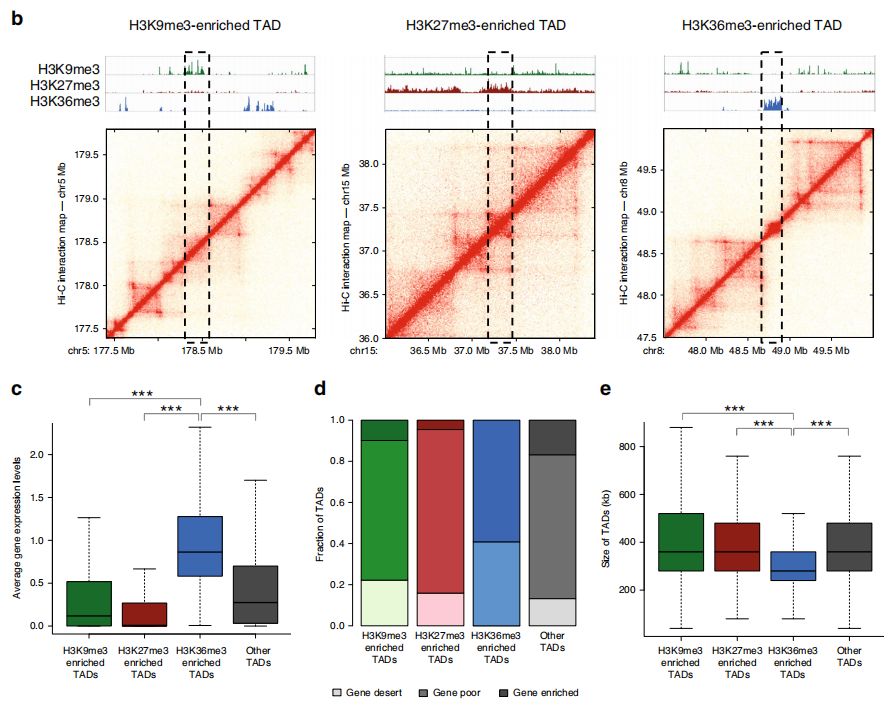
Fig. 2 富含組蛋白標記的TAD亞群鑒定
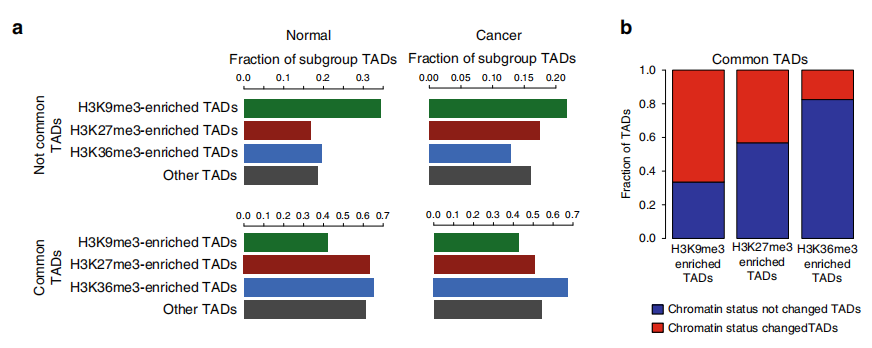
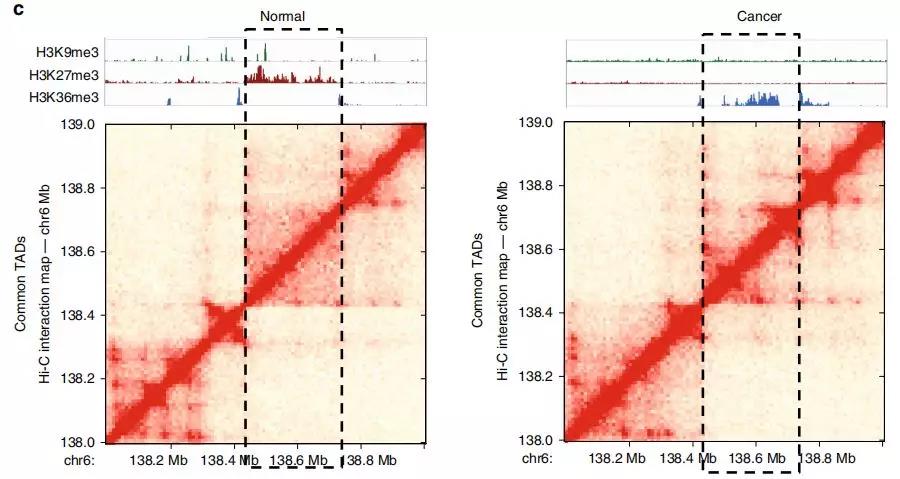
Fig. 3前列腺癌細胞中改變表觀遺傳狀態的TADs的鑒定。
3、前列腺癌細胞的增強子-啟動子環
作者鑒定了正常細胞和前列腺癌細胞中染色質內的顯著的loops,發現?70%的啟動子和增強子區域以及?90%的CTCF峰與染色質相互作用錨重疊。與正常的增強子-啟動子環調控的基因相比,癌癥特異性增強子-啟動子環調控的基因在癌細胞中顯示出更高的基因表達,表明癌癥特異性環有助于增強基因表達。
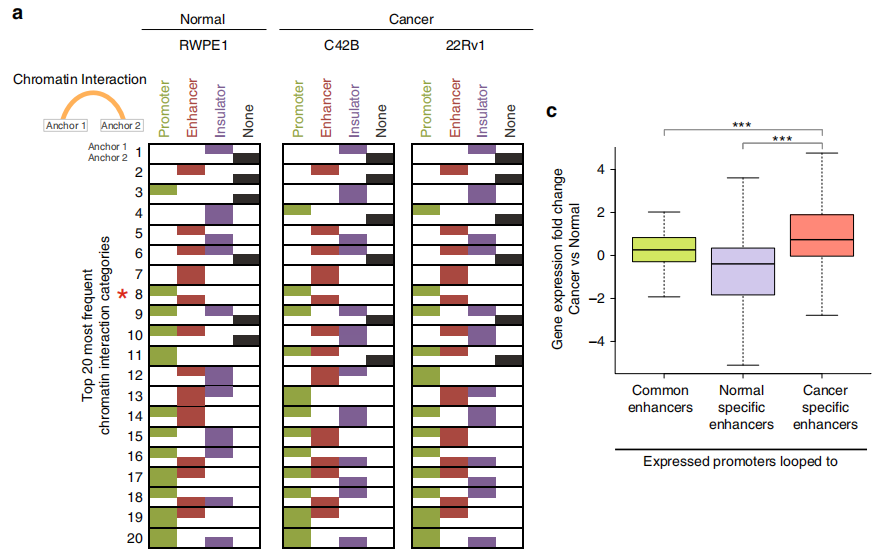
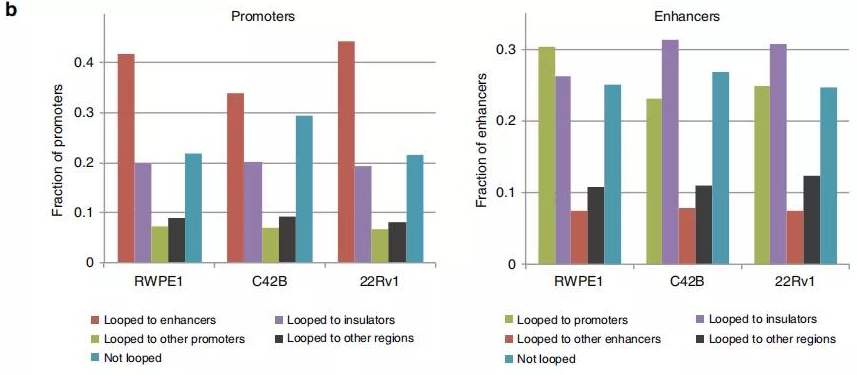
Fig. 4增強子-啟動子環的鑒定
4、細胞類型特異增強子-啟動子環上的TFs
轉錄因子(TFs)是關鍵的調控因子,其通過與DNA調控元件結合,可以導致眾多基因轉錄的變化。細胞類型特異性TFs與調節元件結合和HAT募集后,可產生區分細胞類型的H3K27ac表觀基因組(增強子)圖譜。作者使用NOMe-seq查找NDR,從RWPE1和C42B細胞的全基因組范圍的H3K27ac峰中提取TF結合的基因組位置。結合RNA-seq分析和癌癥基因組圖譜(TCGA)基因表達數據集,作者發現了一個癌癥特異TF基序FOXA1
,在C42B前列腺癌細胞增強子-啟動環中的癌癥特異性增強子的31%由FOXA1結合,它是癌癥轉錄組的主要調節劑。增強子-啟動子環不僅導致FOXA1表達增加,而且上調的FOXA1蛋白還通過介導許多其他的增強子-啟動子環導致基因表達增加,從而助力于癌癥的轉錄組。對啟動子與FOXA1結合的增強子相連的基因研究顯示,參與細胞周期的基因(例如CDK4,CDC23,MYC)以及雄激素受體(AR)和雄激素響應基因(例如GRHL2)是FOXA1結合的目標基因。
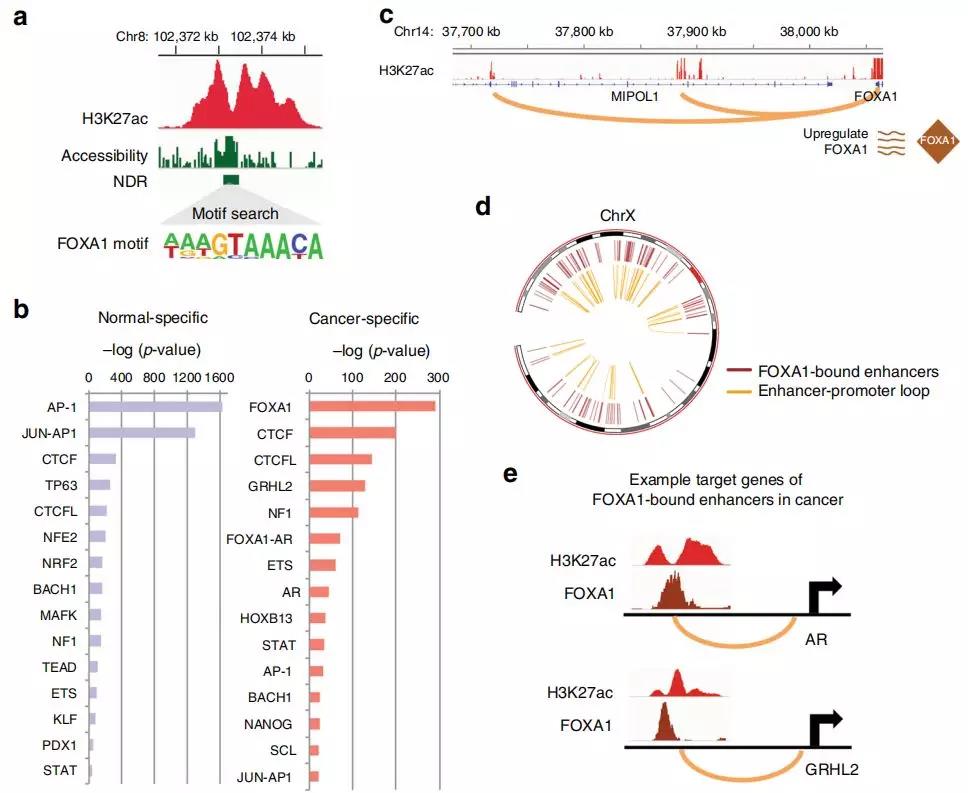
Fig. 5 在細胞類型特異性增強子上富集的TFs與啟動子成環
5、AR基因座周圍的染色質結構
作者發現AR基因的增強子-啟動子環對前列腺癌細胞具有特異性,原位Hi-C證明與RWPE1正常細胞相比,22Rv1前列腺癌細胞增強子-啟動子的相互作用更頻繁。在AR基因附近也有癌癥特異性的小TADs(圖6a)。H3K27ac ChIP-seq圖譜顯示,在22Rv1前列腺癌細胞中,AR基因啟動子所在的TAD中發現的多種增強子具有活性,而在RWPE1正常細胞中則沒有活性(圖6b)。與RWPE1正常細胞相比,癌細胞中更大的染色質接觸頻率可能有助于上調22Rv1前列腺癌細胞中的AR表達。
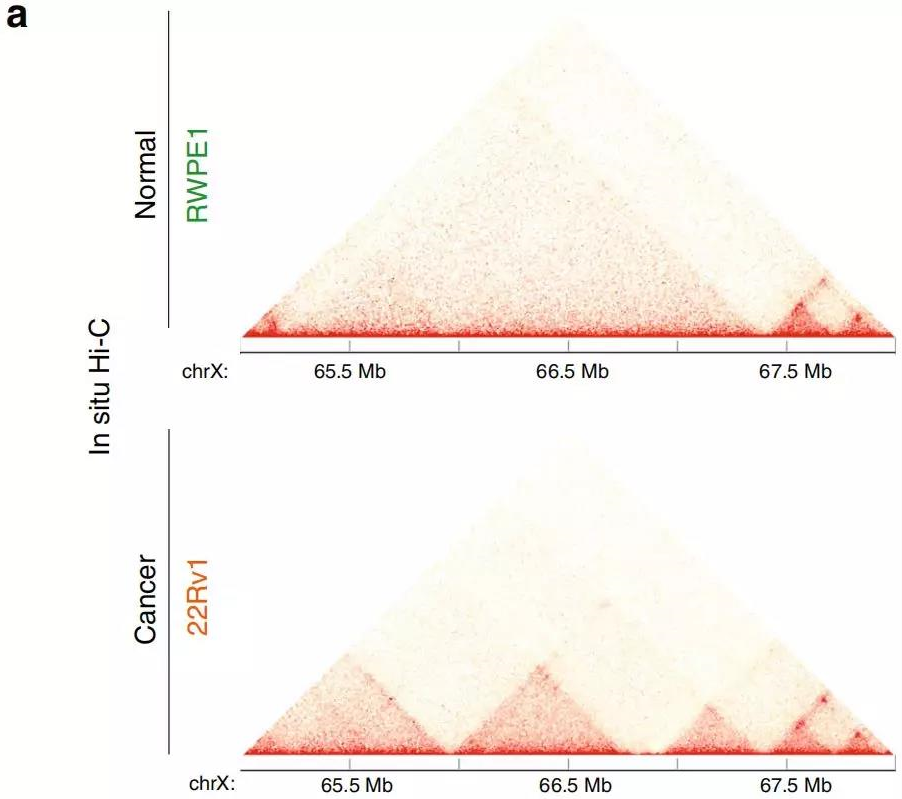
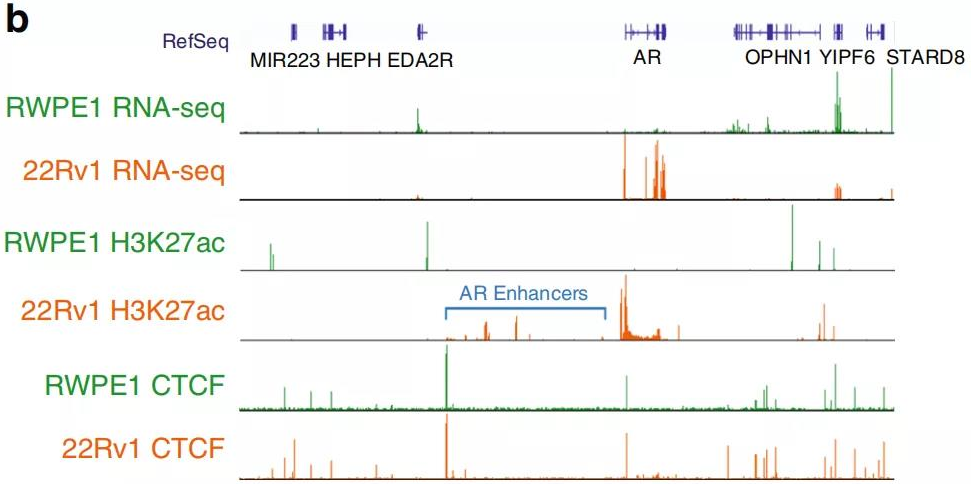
Fig. 6 前列腺癌細胞AR位點周圍染色質結構改變
研究總結:
作者使用原位Hi-C繪制正常細胞和前列腺癌細胞中的高分辨率染色質相互作用圖譜,通過原位Hi-C和ChIP-seq、RNA-seq數據相結合確定正常細胞和前列腺癌細胞TADs的大小和表觀遺傳狀態的改變相關。此外,作者在正常和前列腺癌中,確定了特異性增強子-啟動子環和涉及的轉錄因子。例如,研究顯示FOXA1富含前列腺癌特異性增強子-啟動子環錨;在前列腺癌細胞中,雄激素受體(AR)基因座周圍的染色質結構發生了改變,3D表觀基因組圖譜的創建使人們可以更好地了解前列腺癌生物學和基因調控機制。
金開瑞DLO Hi-C技術優勢:
1. 微量細胞建庫:正常建庫與生信分析的樣本量可低至10萬個核。
2. 高成功率:細胞樣本文庫構建成功率幾乎為100%。
3. 建庫周期短:只需執行兩輪簡單的消化和連接步驟即可獲得高質量的文庫。
4. 數據更準確:測序前質檢,確保數據準確性
5. 分辨率更高:在測序數據量更少的情況下,互作矩陣分辨率更高,染色質結構分析得到的數據也更多
6. 較高的信噪比:使用多種措施來減少噪音,保證高質量的數據輸出,分析更準確。
7. 量身定制個性化分析方案:提供DLO Hi-C的標準分析外,更注重與RNA-Seq、ChIP-Seq、ATAC-Seq和甲基化等多組學表觀遺傳分析,提供個性化的生信分析方案。
下一條:一文輕松讀懂噬菌體展示技術
最新動態
-
09.23
中藥的現代詮釋:外泌體如何革新傳統醫學?
-
07.02
1+1>2!深度解析RNA測序數據挖掘邏輯和后期實驗設計思路,輕松研獲10+ SCI
-
07.01
“稻”亦有道——盤點近期水稻研究的重大突破
-
06.28
科學與美學的結合體:植物亞細胞定位技術詳解
-
06.28
“聚焦新質生產力,激發科研新動能”|LCA躋身蛋白互作研究的新銳力量
-
06.05
知無不“研”|一文讀懂免疫共沉淀技術(Co-IP)
-
05.14
四大研究利器(Co-IP、BIFC、Y2H、GST pull-down)助力速配蛋白互作“最佳拍檔”
-
05.14
高效、精準、直觀、實時——取經“蛋白互作研究翹楚”BIFC!
-
05.14
轉染效率低、干擾效果差、重復性欠佳...siRNA研究頻遇“攔路虎”怎么辦?
-
04.22
一文讀懂EMSA技術核心要點,讓“emsa” 秒變“easy”










