DLO-HiC三維基因組
每個人體內都有著兩米長的DNA,它緊密折疊在直徑10微米小的細胞核內。基因組的三維空間結構對基因組的表達、調控等功能有重要的影響。
基因組三維空間結構與功能的研究簡稱三維基因組學(Three-Dimensional Genomics, 3D Genomics),指在考慮基因組序列、基因結構及其調控元件的同時,對基因組序列在細胞核內的三維空間結構,及其對基因轉錄、復制、修復和調控等生物過程中功能的研究。
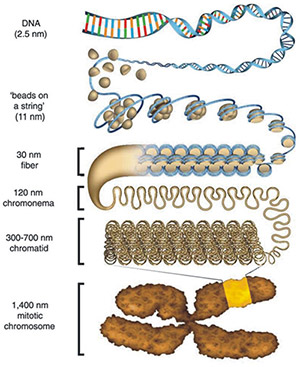
細胞核中染色質折疊模型示意圖
(引自 Ou HD, Phan S, Deerinck TJ, Thor A, Ellisman MH, O'Shea CC. ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells. Science. 2017;357(6349):eaag0025.)
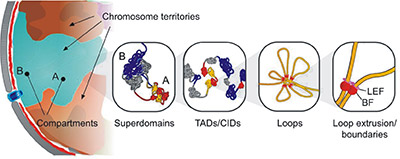
細胞核中染色質三維空間結構分解
(引自 Pueschel R, Coraggio F, Meister P. From single genes to entire genomes: the search for a function of nuclear organization[J]. Development, 2016 , 143 (6) :910.)
2003 年,Job Dekker提出了染色質構象捕獲技術(Chromatin Conformation Capture, 3C),用于測定特定的點到點之間的染色質交互作用。2006年,在3C技術基礎上,Simonis M開發了4C技術(Circularized Chromatin Conformation Capture),用于測定一點到多點之間的染色質交互作用。Dostie等人研發了5C技術(Carbon-Copy Chromatin Conformation Capture),用于測定多點到多點之間的染色質交互作用。
為了能捕獲全基因組范圍的染色質相互作用,Job Dekker 開發出 Hi-C 技術,并將其應用于揭示染色質的三維結構中。Hi-C將線性距離遠、空間結構近的DNA片段進行交聯, 并富集交聯的DNA片段進行高通量測序,通過測序數據分析揭示染色質的遠程相互作用,推導基因組的三維空間結構和可能的基因間的調控關系。
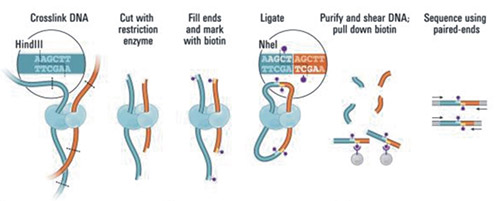
Hi-C 實驗流程示意圖
(引自 Lieberman-Aiden E, van Berkum NL, et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome[J]. Science. 2009, 326(5950):289-93.)
Hi-C 技術可用于研究:1.染色質的層級結構(A/B compartments、TADs、Loops);2. 關聯基因組上原本分散的遠距離調控元件與其具體調控區域;3. 解析基因的轉錄調控、增強子與啟動子的作用機制;4. 協助發現疾病易感位點、DNA 損傷修復、基因組結構變異和表觀遺傳。
武漢金開瑞生物工程有限公司技術顧問、華中農業大學教授曹罡課題組與華中農業大學李國亮教授課題組在國際著名期刊Nature Genetic( IF=38.33 )上聯合發表題為“Digestion-ligation-only Hi-C is an efficient and cost-effective method for chromosome conformation capture”的研究論文。該論文主要介紹了一種新的染色質構象捕獲技術(Digestion-ligation-only Hi-C, DLO Hi-C),此技術信噪比高,能在早期進行質量控制,為解析基因組三維結構提供了一種新型、高效、經濟的研究方法。
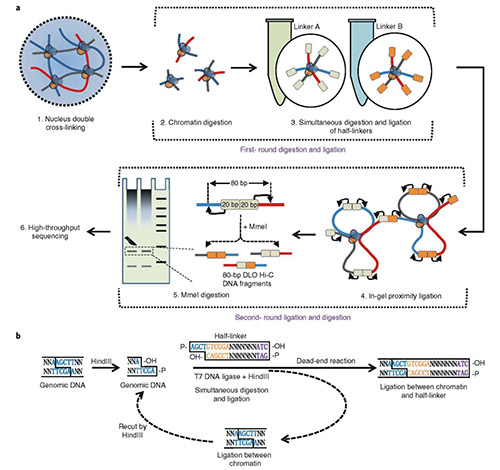
DLO Hi-C 染色質構象捕獲技術設計了巧妙的酶切位點,采用同時酶切酶連的方式,將 DNA 接頭連接在染色質內切酶切口末端上,然后進行鄰近酶連,最后再用 MmeI 內切酶酶切消化,回收固定大小互作 DNA 片段。
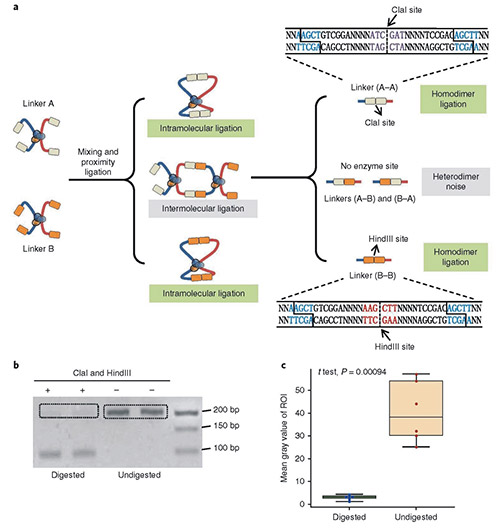
消化后的染色質與兩種半接頭(接頭 A 或接頭 B)連接,如果發生分子內連接,那么接頭連接后(A-A/B-B)就會形成HindⅢ或ClaI酶切位點,而隨機的分子間連接形成的噪音異二聚體(A-B/B-A)上沒有限制酶切割位點。
DLO Hi-C 染色質構象捕獲技術,具有以下優點:
1. 圖譜分辨率高于傳統 Hi-C 技術、DNase Hi-C 技術;
2. DLO Hi-C 簡化 Hi-C 復雜的文庫構建過程;
3. A/B compartments 數據、TADs 數據和 Loops 數據較優于其他 Hi-C 的衍生技術。
RUNX1-mediated alphaherpesvirus-host trans-species chromatin interaction promotes viral transcription. 期刊:Sci Adv. IF:14.14 發表時間:2021
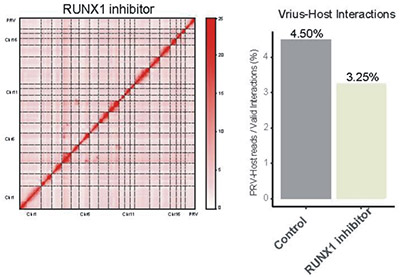
使用偽狂犬病病毒 (PRV) 作為模型病毒,對感染后豬腎PK15細胞系進行了染色體構象捕獲測定,以證明病毒和宿主之間的全基因組特異性跨物種染色質相互作用。發現了PRV 基因組由宿主 DNA 結合蛋白 RUNX1 傳遞到開放染色質和活躍轉錄區。本研究使用了4C、DLO Hi-C、ChIP-seq、ATAC-seq、RNA-seq、DNA FISH等多種技術。
3D genome architecture coordinates trans and cis regulation of differentially expressed ear and tassel genes in maize. 期刊:Genome Biol. IF:10.81 發表時間:2020
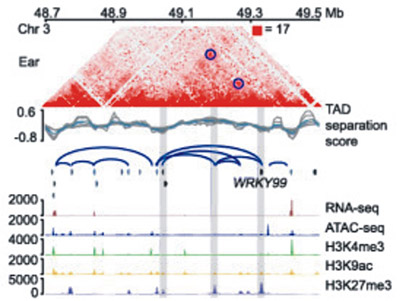
使用 ATAC-seq 和ChIP-seq在玉米穗和流蘇發育過程中識別出56,055和52,633個開放染色質區域(OCR),進一步使用DLO Hi-C在相應組織中以高分辨率系統地繪制染色質相互作用。連接OCR和基因的廣泛染色質環為負責穗和流蘇特異性基因表達的順式和反式調節元件提供了3D視圖。
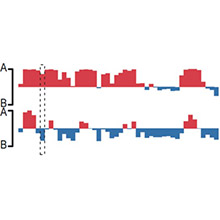

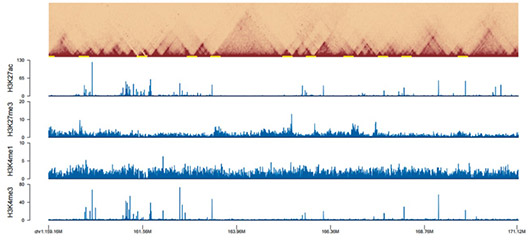
Hi-C與組蛋白修飾ChIP-seq聯合分析
| 項目 | 內容 |
| 建庫、測序 | 每個樣本生成2個文庫,每個文庫120GB測序量,每個樣品產生不少于200GB raw data |
| 標準分析 | 質量控制、染色體順式、反式互作,全基因組交互、A/B compartment分析、TAD分析 |
| 樣本組間差異A/B、TADs、Loops比對 | 商議 |
| Hi-C與RNA聯合分析 | |
| 與其他組學聯合分析 |
最新動態
-
09.23
中藥的現代詮釋:外泌體如何革新傳統醫學?
-
07.02
1+1>2!深度解析RNA測序數據挖掘邏輯和后期實驗設計思路,輕松研獲10+ SCI
-
07.01
“稻”亦有道——盤點近期水稻研究的重大突破
-
06.28
科學與美學的結合體:植物亞細胞定位技術詳解
-
06.28
“聚焦新質生產力,激發科研新動能”|LCA躋身蛋白互作研究的新銳力量
-
06.05
知無不“研”|一文讀懂免疫共沉淀技術(Co-IP)
-
05.14
四大研究利器(Co-IP、BIFC、Y2H、GST pull-down)助力速配蛋白互作“最佳拍檔”
-
05.14
高效、精準、直觀、實時——取經“蛋白互作研究翹楚”BIFC!
-
05.14
轉染效率低、干擾效果差、重復性欠佳...siRNA研究頻遇“攔路虎”怎么辦?
-
04.22
一文讀懂EMSA技術核心要點,讓“emsa” 秒變“easy”










