HI-C技術(三維基因組)經典文獻解讀
信息來源:金開瑞 作者:genecreate 發布時間:2018-10-24 17:29:11
題目:Genome organization and chromatin analysis identify transcriptional downregulation of insulin-like growth factor signaling as a hallmark of aging in developing B cells
通過基因組組織和染色質結構分析確定了胰島素樣生長因子信號的轉錄下調是B細胞開始衰老的標志
期刊:Genome Biology
影響因子:13.214
研究背景
衰老的特征是適應性免疫系統功能的喪失,但其根本原因卻鮮為人知。為了評估年齡對B細胞發育的影響,作者對組蛋白修飾和染色體構象相關的基因表達和染色質特征進行了分析,使用的實驗材料來自不同年齡階段的小鼠的pro-B和pre-B細胞。
研究內容及結果
1. 在衰老過程中,平均pro-B和 pre-B細胞數量分析
使用多參數流式細胞分析小鼠骨髓B細胞,發現老年小鼠的pre-B細胞發生實質性的變化(圖1),老年的(三個月)與年輕的老鼠(約19 - 22日)相比,pro-B細胞大約減少了兩個(圖1 c)、pre-B細胞卻有著近兩倍數量的減少(圖1 d),該結果與先前的報道一致。與此相反的是,發現在老年小鼠骨髓中會重新循環成熟的B細胞(圖1e),不能排除被分類的衰老pro-B細胞群體中存在少量成熟B細胞污染。可能是由于成熟B細胞再循環數量增加,以及在衰老的骨髓中,B細胞前體(IgM-)和成熟細胞(IgM+)分離不那么明顯。
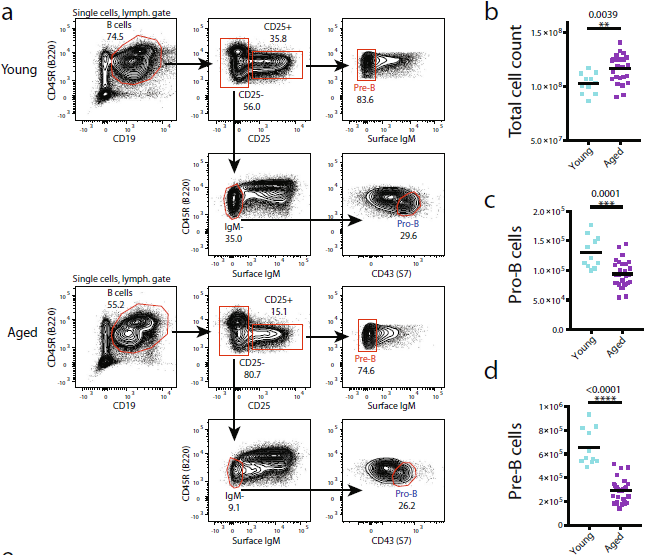
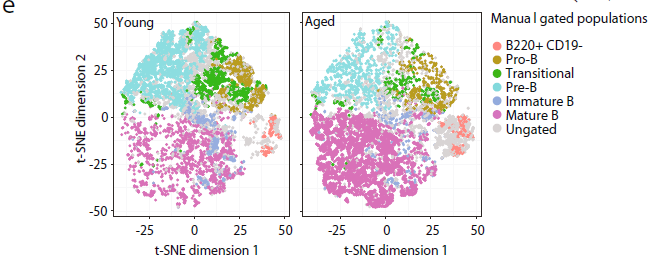
圖1 衰老小鼠B細胞前體基因表達變化
2. pro-B和pre-B 細胞中的基因表達分析
對核糖體RNA (rRNA)耗盡的總RNA轉錄組分析發現,衰老pro-B細胞中有82個顯著上調和54個下調基因,衰老pre-B細胞中有23個上調和33個下調基因(圖2 a , b)。pro-B細胞和pre-B細胞之間的年齡特異性表達變化是相關的,即使只有一種細胞類型也能達到顯著性(圖2 c)。在pro-B和pre-B細胞階段的老化過程中,一些基因被降低了,這些基因編碼胰島素樣生長因子(IGF)信號傳導途徑的組成部分,如IRS1和IGF1r。IRS1是通路的一個關鍵組成部分,被發現其是衰老pre-B細胞信使RNA (mRNA)水平上最顯著的下調基因。因此,作者研究了IRS1蛋白水平,發現在pro-B細胞和pre-B細胞的老化過程中,蛋白含量都有所下降,這表明IRS1介導的特定年齡mRNA變化會降低蛋白質水平(圖2d)。對于衰老前B細胞中下調的基因,在32個啟動子中,有19個啟動子的活性啟動子或活性調控區域狀態被降低。與大多數上調基因的觀察結果相似,這些變化大多是細微的,與這些基因表達中相對較小的折疊變化相一致(圖2b)。
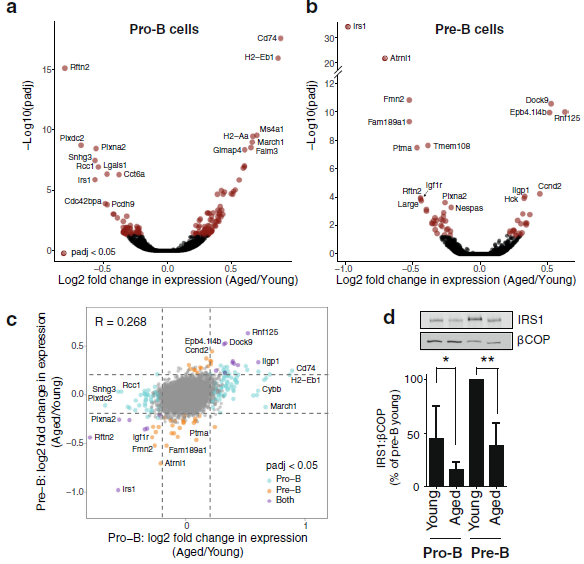
圖2 基因表達在pro-B和pre-B 細胞隨著年齡的老化而改變
3. 初期的pro-B 和pre-B細胞的RNA和microrna表達分析
總RNA水平既反映了轉錄速率,也反映了RNA穩定性等下游過程。為了更直接地探討衰老對基因轉錄的影響,作者從年輕和衰老的pro-B細胞中分離出核RNA,以豐富新生轉錄本,并以內含子轉錄的整體變化作為新生轉錄的一種具體衡量指標。同時,在總RNA中檢測到17%(175個中有30個)的DEGs在核RNA測序(RNA-seq)分析中也是DEGs,并且整體的折疊變化是相關的,從而可以認為更多的基因存在與年齡相關的顯著性差異轉錄(圖3a)。值得注意的是,一些年齡下調基因,如Plxdc2、Igf1、Igf2bp3和Igf1r,以及上調的基因,如Adam19和Tmem163,都與IGF信號或2型糖尿病有關。更廣泛地說,DEGs的KEGG 通路分析突出了幾個與營養信號相關的代謝通路。
通過核RNA-seq分析顯示Nespas是衰老pro-B細胞中最顯著的下調基因之一(圖3a)。Nespas是非編碼轉錄本,參與調控印跡,是miRNAs miR-296和miR-298的非編碼前體RNA。為了探索B細胞前體中miRNA表達與衰老的關系,作者進行了小RNA-seq,在pro-B細胞和pre-B細胞中鑒定出34個有差異顯著的mirna(圖3b、c)。其中20.6%在pro-B細胞和pre-B細胞中均表現出表達差異。該分析證實了miR-296和miR-298的顯著下調,與新生RNA-seq檢測到的Nespas水平變化一致。作者還觀察到7個Let-7家族基因和miR-223在pro-B和pre-B細胞衰老時上調(圖3b, c)。不同表達的microrna在衰老過程中被分離成簇,表達變化相似,并且其中一些表達在pro-B細胞和pre-B細胞之間也有調節作用(圖3c)。
Let-7 mirna由12個具有相同序列的成員組成,分別從基因組位點Let-7a1、Let-7a2、Let-7b、Let -7c、Let -7c、Let-7d、Let-7e、Let-7f1、Let-7f2、Let-7g、Let-7i和miR-98進行表達。Let-7b和7 c在年老的pre-B細胞顯著上調microrna,作者檢查了核RNA-seq數據,年老的pro-B細胞非編碼RNA 在15號染色體上顯著增加。因此推測,這個轉錄組可能是這些microrna的RNA前體。
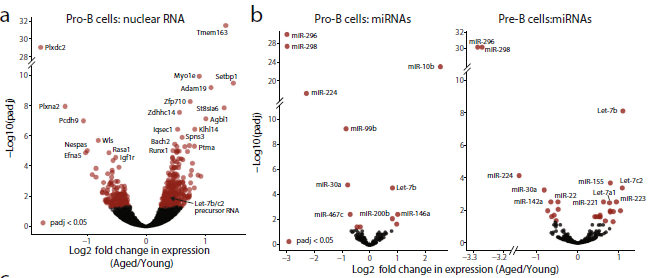

圖3 初期的pro-B和pre-B細胞的RNA和microrna表達隨著年齡的老化而改變
4. 染色質可及性和pre-B細胞中的H3K27me3與年齡相關的變化分析
在老年pre-B細胞中,發現10個可及性明顯較低的區域和4個可及性較高的區域(圖4a)。可及性低的兩個區域與Irs1啟動子區域重疊,另一個區域與Irs1轉錄終止位點接近(圖4a);對于組蛋白修飾物H3K27me3,只確定了兩個峰,在年輕和衰老pre-B細胞之間存在顯著的富集差異(圖4b),第一個峰位于Irs1啟動子上,在衰老pre-B細胞中表現出顯著的富集,與Irs1轉錄下調一致,第2個峰位于15號染色體上Let-7b/c2調控區域(圖4b)。
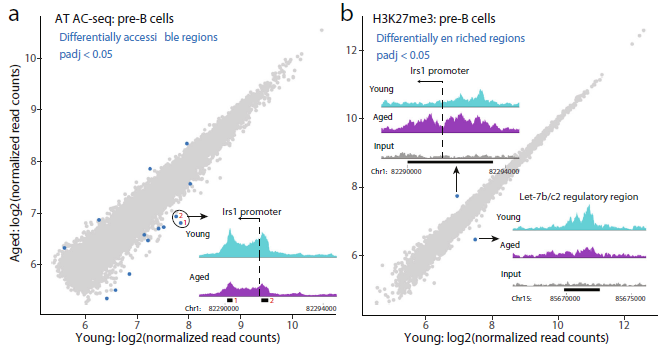
圖4 染色質可及性和pre-B細胞中H3K27me3與年齡相關的變化分析
5. 基因表達的變化與老化的B細胞前體染色質變化分析
如圖5所示,基于閾值方法識別高置信度位點,顯示出染色質可及性的變化和組蛋白在衰老過程中的修飾變化非常少,僅顯示了Irs1和Let-7 mirna轉錄調控的重要染色質成分。這表明染色質隨著年齡變化而的變化,具體體現在H3K4me3和H3K27me3的占有率,這一現象可能對基因表達有影響,但進一步研究發現,如果有基因表達發生了變化,也會引起相關的染色質發生變化。值得注意的是,H3K4me3和H3K27ac更符合差異基因表達而不是染色質可及性。
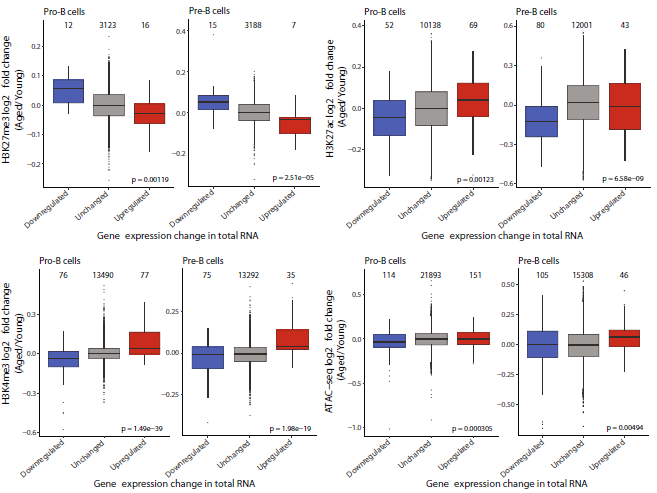
圖5 基因表達的變化與老化的B細胞前體染色質變化
6. 在pre-B細胞中進行染色質狀態分析,識別與年齡相關的變化
基于平衡類內同質性和類間異質性,作者選擇了一個的16狀態模型(圖6a)。該模型分為6大類,稱為“多梳”、“雙價”、“活性啟動子”、“活躍的調節區域”、“絕緣體”和“背景”。為了整合這些變化,并確定在染色質狀態下啟動子表現出類似轉變的基因組,作者使用無聚類法。將啟動器分隔為九個顯示不同狀態變化模式的集群,這些基因也表現出與染色質狀態改變相一致的基因表達調控機制。例如,從主動啟動子狀態到雙價或多梳狀態的基因表達,通常在衰老的時候會減少。相反,顯示相反轉變的基因(聚類6)或其多梳狀態占用減少的基因(聚類5)傾向于表達增加(圖6 c)。
作者接下來評估了所有基因啟動子的染色質狀態變化,這些基因在pre-B細胞中表現出明顯的年齡特異性表達變化。年齡上調基因的啟動子只出現了細微的變化,也有少數例外,包括新發現的Let-7b/c2前體基因、Ccnd2 (cyclin D2)和Mctp1。啟動子區域從年輕時的二價顯性狀態轉變為年老的pre-B細胞更活躍狀態(圖6d)。在全基因組分析中,Ccnd2和Mctp1都被分配到6組(圖6c)。另外觀察到Irs1啟動子上發生了實質性的變化,隨著年齡的增長,活躍狀態轉變為二價狀態(圖6d),在全基因組分析中也發現了這一點,在集群2中分離(圖6c)。因此,染色質狀態分析顯示了在啟動子上染色質改變的基因。
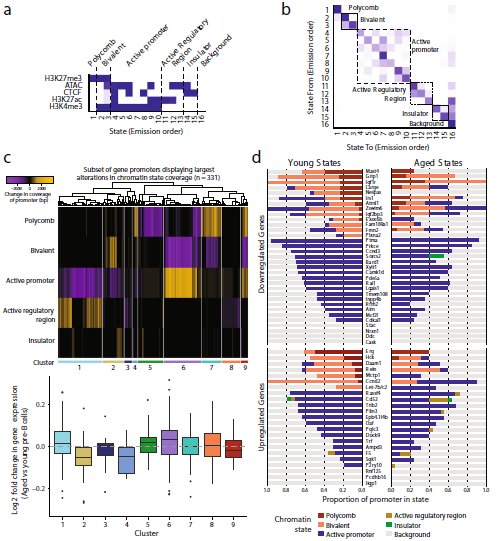
圖6 pre-B細胞中染色質狀態分析
7. pre-B細胞的基因組組織變化
衰老與基因組組織的變化有關,為了研究B細胞發育過程中是否會發生這種變化,以及是否與基因調控的變化相關,作者首先在B細胞前期分離的細胞核中進行了Hi-C檢測。利用主成分分析(PCA)和H3K4me3數據,使用Hi-C數據將基因組分為A(活躍的)和B(壓抑的)兩部分。這一分析表明,年輕和老年pre-B細胞之間的染色體間隔幾乎相同。在9928個區域中,有100個區域在老化后的隔間得分有顯著變化(圖7a,左上),40個區域的隔間得分顯著增加,其中3個區域從壓抑的B隔間(負分)變為活躍的A隔間(正分)。相反,60個區域的間隔分數顯著降低,有6個從A隔間切換到B隔間。值得注意的是,A-to-B轉換區域之一包含1號染色體上的Irs1基因(圖7a,下),間隔評分降低的區域與10個基因重疊,這些基因在衰老pre-B細胞中表達顯著降低,其中包括Plxna2和Igf1r。另外,發現隨著隔間分數的增加基因表達呈高度顯著的增加趨勢,反之亦然(圖7a,右上方)。這表明,在活躍和壓抑的核環境之間基因的重新定位可能有助于在衰老過程中調節基因表達。
為了獲得pre-B細胞前期啟動子相互作用及其衰老動力學的高分辨率圖像,作者進行了啟動子捕獲Hi-C(PCHi-C),對年輕和老年小鼠的細胞進行研究,并利用CHiCAGO試驗確定具有統計學意義的啟動子相互作用。為了區分這種相互作用,作者分析了啟動子PIR相互作用。與其他細胞類型的PCHi-C結果一致,發現PIRs富集于與基因調控相關的特征,包括H3K4me3、H3K27me3和H3K27ac(圖7b)。這表明,許多檢測到的物理交互作用將啟動子與調控元件連接起來。上調基因的啟動子在衰老pre-B細胞中獲得了與其他啟動子的相互作用,并且很少與啟動子或PIRs失去相互作用,而下調基因在衰老過程中經常與兩者失去相互作用(圖7c)。隨后分析CHiCAGO互動分數的變化,這顯示了上調基因分數的普遍趨勢,存在更強或更頻繁的相互作用,而下調基因的得分較低,提示與調控元件失去聯系(圖7d)。總的來說,作者沒有發現基因表達變化與衰老后PIRs染色質改變之間有顯著的聯系(圖7e),一些被下調的基因失去了與活躍區域的相互作用,這一趨勢沒有達到顯著性(圖7f)。

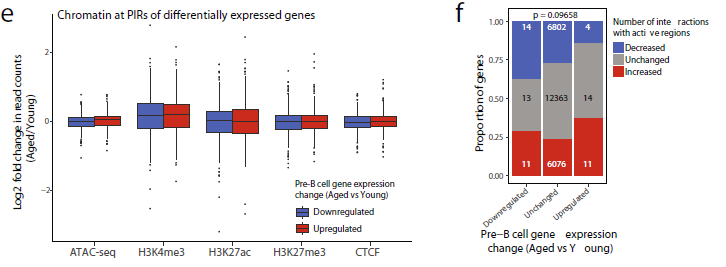
圖7 B細胞前體基因組組織的年齡特異性變化
8. 衰老pre-B細胞啟動子相互作用的重新連接分析
作者接下來探討了不同年齡的基因表達變化與PIRs染色質狀態變化之間的關系。在某些情況下,例如在P2ry10、Epb4.1l4b和Ccnd3基因上,上調或下調基因的PIR分別顯示活躍染色質標記的水平略有增加或減少(圖8b)。作者還觀察了一些上調基因啟動子與染色質活躍區(包括啟動子和PIRs)連接的案例,值得注意的是,這種相互作用的“重新連接”的例子包括隨年齡下降的Irs1基因,它在衰老細胞中丟失了一些相互作用,例如與Cul3啟動子周圍高度活躍的染色質的相互作用(圖8a)。這些結果表明,在衰老過程中,即使啟動子的表達發生了改變,染色質在PIRs上的調節并不經常發生。隨著年齡的增長,基因表達的變化通常與特定啟動子相互作用的“重新連接”有關。這可能有助于在活躍(A)和非活躍(B)基因組環境之間進行更廣泛的轉換。
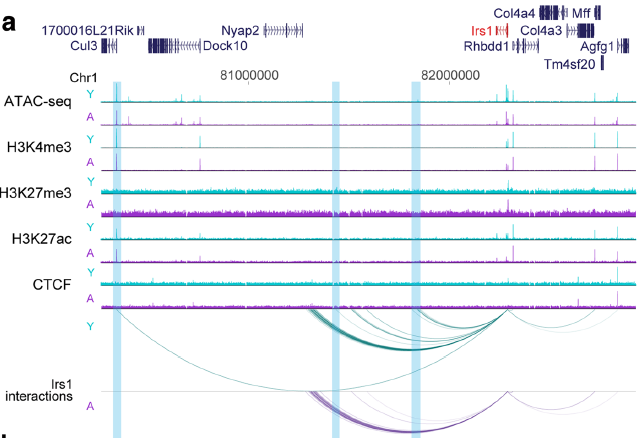

圖8衰老pre-B細胞啟動子相互作用的重新連接
文章小結
與幼齡鼠相比,老年小鼠的大多數基因的表達水平通常被保存在pre-B細胞中。但是,許多基因表達會有年齡特異性的變化,包括microRNA編碼基因。這些變化是由染色質結構的多層改變所引起的,包括染色質的可及性、組蛋白修飾、遠程啟動子互動和核區劃分。先前的研究表明,年齡差異化與促進調節元素交互的變化有關。作者發現,在衰老B細胞前體上,會重新連接原有的差異化與促進調節元素相互作用。最終,作者確定了胰島素樣生長因子(IGF)信號通路的轉錄負調控機制,特別是對Irs1和Let-7 microRNA表達的負轉錄調控,這是一項重要的老年表型特征。這些變化與H3K27me3的具體改變有關,這表明,多細胞介導的抑制在pre-B老化中起著重要作用。
解析文獻
Hashem Koohy, Daniel J, et al. Genome organization and chromatin analysis identify transcriptional downregulation of insulin-like growth factor signaling as a hallmark of aging in developing B cells. Genome Biology , 2018 , 19 (1) :126
參考文獻
1. Imakaev M, Fudenberg G, et al. Iterative correction of Hi-C data reveals hallmarks of chromosome organization. Nat Meth. 2012;9:999–1003.
2. Wingett S, Ewels P, et al. HiCUP: pipeline for mapping and processing Hi-C data. F1000Res. 2015;4:1310.
3. Mifsud B, Tavares-Cadete F, et al. Mapping long-range promoter contacts in human cells with highresolution capture Hi-C. Nat Genet. 2015;47:598–606.
4. Cairns J, Freire-Pritchett P, et al. CHiCAGO: robust detection of DNA looping interactions in Capture Hi-C data. Genome Biol. 2016;17:127.
最新動態
-
09.23
中藥的現代詮釋:外泌體如何革新傳統醫學?
-
07.02
1+1>2!深度解析RNA測序數據挖掘邏輯和后期實驗設計思路,輕松研獲10+ SCI
-
07.01
“稻”亦有道——盤點近期水稻研究的重大突破
-
06.28
科學與美學的結合體:植物亞細胞定位技術詳解
-
06.28
“聚焦新質生產力,激發科研新動能”|LCA躋身蛋白互作研究的新銳力量
-
06.05
知無不“研”|一文讀懂免疫共沉淀技術(Co-IP)
-
05.14
四大研究利器(Co-IP、BIFC、Y2H、GST pull-down)助力速配蛋白互作“最佳拍檔”
-
05.14
高效、精準、直觀、實時——取經“蛋白互作研究翹楚”BIFC!
-
05.14
轉染效率低、干擾效果差、重復性欠佳...siRNA研究頻遇“攔路虎”怎么辦?
-
04.22
一文讀懂EMSA技術核心要點,讓“emsa” 秒變“easy”










