ATAC-seq技術簡介及與ChIP-Seq的異同
信息來源:金開瑞 作者:genecreate 發布時間:2019-07-17 09:30:31
ATAC-Seq簡介
ATAC-seq全稱Assay for Transposase Accessible Chromatin with high-throughput sequencing,即利用轉座酶研究染色質可進入性的高通量測序技術。
要理解這個東西有什么用,首先得認識一下染色體/質的結構。
真核生物的核DNA并不是裸露的,而是有蛋白質即組蛋白與之相結合的。DNA一圈一圈得纏繞在組蛋白上,形成串珠式的結構。而這樣的結構還能夠進一步折疊、濃聚,并在其他架構蛋白的輔助下,形成染色體。
這樣做的意義在于,將超長的DNA鏈,折疊成很小很小的結構,從而能夠塞進小小的細胞核里。比如說,人類的DNA鏈如果完整展開,那么大概會有2米那么長。而經過這樣的折疊,就變成了納米至微米級染色體/質的結構了。
但是我們又知道,DNA的復制,基因的轉錄,是需要將DNA的高級結構解開的。這就類似于我們要從zip文件中獲得信息的話,首先需要解壓縮。但是,這里的解壓縮并不需要將整個zip全部打開,而只需要打開一部分,即需要表達基因的區域即可。
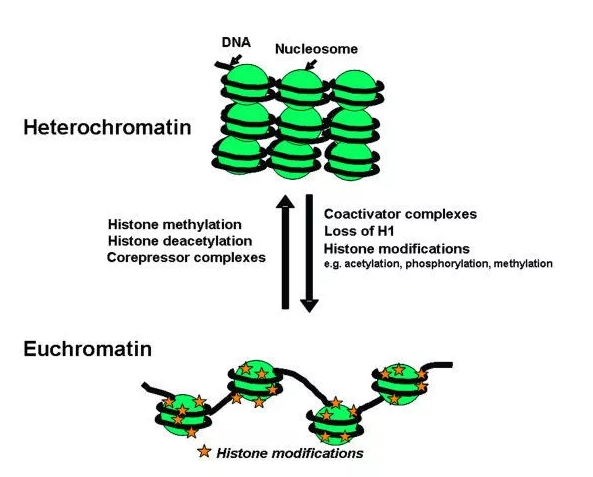
這部分打開的染色質,就叫開放染色質(open chromatin)。而打開的染色質,就允許一些調控蛋白(比如轉錄因子和輔因子)跑過來與之相結合。而染色質的這種特性,就叫做染色質的可進入性(chromatin accessibility)。
我們如何去尋找開放的染色質區域呢?
傳統的實驗方法主要是借助MNase-seq和DNase I hypersensitivity assay。
這兩個實驗的主要思路是一致的:染色質變得開放,就意味著DNA和組蛋白的濃聚程度降低,就會有一部分DNA暴露出來。而一旦失去了蛋白質的保護,這部分DNA就可以被DNA酶(MNase或DNase I)所切割。然后,我們再把切割完的DNA拿來測序,和已知的全基因組序列相比較,就能發現被切掉的是哪些東西,沒有被切掉的地方又在哪里,就知道開放的染色質區域在哪里了。
不過,這兩個方法有明顯的缺陷。即耗時費力與重復性差。具體就不展開講,親手做過的就知道這兩個方法有多么討厭。
2013年,美國Stanford大學的William Greenleaf教授研發了一種全新的方法,利用DNA轉座酶結合高通量測序技術,來研究染色體的可進入性,即ATAC-seq。
DNA轉座,是一種把DNA序列從染色體的一個區域搬運到另外一個區域的現象,由DNA轉座酶來實現。這種轉座插入DNA,也是需要插入位點的染色質是開放的,否則就會被一大坨高級結構給卡住。那么,我們只要人為地,將攜帶已知DNA序列標簽的轉座復合物(即帶著測序標簽的轉座酶),加入到細胞核中,再利用已知序列的標簽進行PCR后測序,就知道哪些區域是開放染色質了。而這也就是ATAC-seq的原理。
ATAC-seq出來的結果,和傳統方法出來的結果具有很強的一致性,同時也和基于組蛋白修飾marker的ChIP-seq有較高的吻合程度。而相比起來,ATAC-seq的重復性,比MNase-seq和DNase-seq的更強,操作起來也更加簡單,而且只需要很少的細胞/組織量,同時出來的信號更加漂亮。目前已經是研究染色質開放性首選的技術方法。
ATAC-Seq與CHIP-Seq的異同
ATAC-Seq與ChIP-Seq的不同的是ATAC-Seq是全基因組范圍內檢測染色質的開放程度,可以得到全基因組范圍內的蛋白質可能結合的位點信息,一般用于不知道特定的轉錄因子,用此方法與其他方法結合篩查感興趣的特定調控因子;但是ChIP-Seq是明確知道感興趣的轉錄因子是什么,根據感興趣的轉錄因子設計抗體去做ChIP實驗拉DNA,驗證感興趣的轉錄因子是否與DNA存在相互作用。ATAC-Seq、ChIP-Seq、Dnase-Seq、MNase-Seq、FAIRE-Seq整體的分析思路一致,找到富集區域,對富集區域進行功能分析。
1. ChIP-Seq是揭示特定轉錄因子或蛋白復合物的結合區域,實際是研究DNA和蛋白質的相互作用,利用抗體將蛋白質和DNA一起富集,并對富集到的DNA進行測序。
2. DNase-Seq、ATAC-Seq、FAIRE-Seq都是用來研究開放染色質區域。DNase-Seq是用的DNase I內切酶識別開放染色質區域,而ATAC-seq是用的Tn5轉座酶,隨后進行富集和擴增;FAIRE-Seq是先進行超聲裂解,然后用酚-氯仿富集。
3. MNase-Seq是用來鑒定核小體區域。
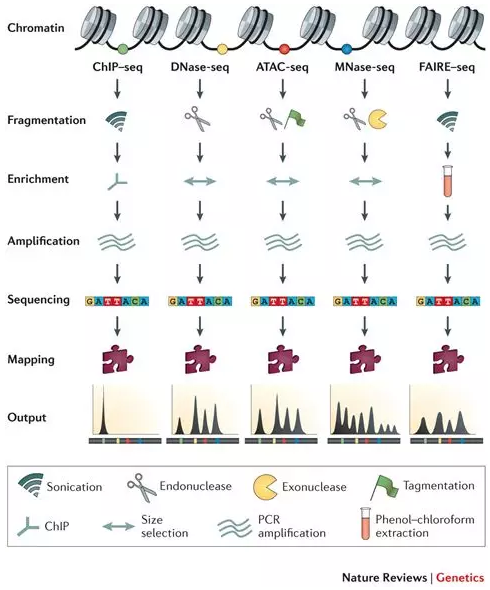
An overview of ChIP–seq, DNase-seq, ATAC-seq,
MNase-seq and FAIRE–seq experiments
最新動態
-
09.23
中藥的現代詮釋:外泌體如何革新傳統醫學?
-
07.02
1+1>2!深度解析RNA測序數據挖掘邏輯和后期實驗設計思路,輕松研獲10+ SCI
-
07.01
“稻”亦有道——盤點近期水稻研究的重大突破
-
06.28
科學與美學的結合體:植物亞細胞定位技術詳解
-
06.28
“聚焦新質生產力,激發科研新動能”|LCA躋身蛋白互作研究的新銳力量
-
06.05
知無不“研”|一文讀懂免疫共沉淀技術(Co-IP)
-
05.14
四大研究利器(Co-IP、BIFC、Y2H、GST pull-down)助力速配蛋白互作“最佳拍檔”
-
05.14
高效、精準、直觀、實時——取經“蛋白互作研究翹楚”BIFC!
-
05.14
轉染效率低、干擾效果差、重復性欠佳...siRNA研究頻遇“攔路虎”怎么辦?
-
04.22
一文讀懂EMSA技術核心要點,讓“emsa” 秒變“easy”










