非編碼RNA技術總結
信息來源:金開瑞 作者:genecreate 發布時間:2019-01-04 09:59:14
長鏈非編碼RNA(long noncoding RNA,lncRNA)是一類長度>200bp的RNA,由RNA聚合酶Ⅱ轉錄,lncRNA具有保守的二級結構,大部分不編碼蛋白質。LncRNA發揮功能的方式很廣,可以與蛋白、DNA和RNA相互作用,參與多種生物學過程的調控。主要包括基因組表觀遺傳修飾、調控轉錄后翻譯、發揮ceRNA、增強子RNA作用等,從而對細胞的增殖、分化、遷移、凋亡、免疫等發揮調控作用。
非編碼RNA可火了,miRNA大火燒了十幾年,現在慢慢地把lncRNA的領域也給點著了。現在可能連實驗室的插槍頭阿姨都知道絕大多數的RNA分子并不翻譯,這非編碼RNA(noncoding RNA),包括miRNA、lncRNA等。
目前主要研究方向集中在非編碼RNA 及其相互結合的DNA、RNA、蛋白質等科學問題,而科學問題的解答依賴于實驗技術的創新使用和革命。
IncRNA可以海陸空全面調控細胞功能,厲害了,其實主要指lncRNA,而miRNA、siRNA主要作用于mRNA層面,本文按非編碼RNA的三個調控維度,就當前主要的實驗技術簡單介紹一下。
(一)DNA維度
1)作用的ncRNA類型:lncRNA等
2)研究方法介紹有:
和DNA序列結合的調控目的無非是①阻止下游基因的轉錄②把轉錄因子、甲基化酶等招惹過來修飾DNA序列。他們的共同特點是和解鏈的DNA的某一條鏈結合,那么研究方法包括

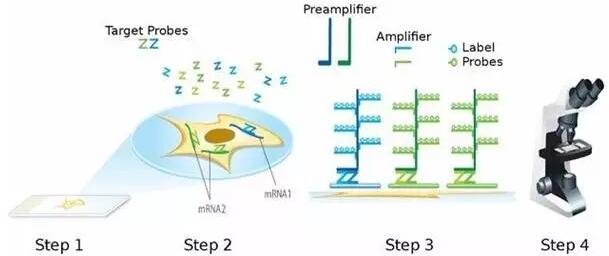
1. RNA原位雜交(RNAin situ hybridization)
檢測該ncRNA是否位于核內,如果不在核內,那就不用考慮和DNA作用了。
RNA原位雜交是指將特定標記的已知順序核酸為探針與細胞或組織切片中核酸進行雜交,從而對特定核酸順序進行精確定量定位的過程。原位雜交可以在細胞標本或組織標本上進行。根據RNA序列設計cDNA、cRNA探針,對目標進行定位。
2. ChIRP(chromatin isolation by RNA purifications)
是一項通過純化RNA 分子從而獲得其結合的染色質片段( 包括RNA 結合蛋白與基因組DNA) 的實驗方法。
大概流程:首先細胞交聯,使lncRNA、染色質及被固定連接;隨后通過超聲將染色質打斷,并使用帶有生物素標記的tiling oligos與長鏈非編碼RNA 分子雜交;之后利用親和素磁珠純化長鏈非編碼RNA 及其結合的染色質片段;后續分析和測序。

3. ncRNA-DNA三聯體的體外同位素檢測
為了進一步檢驗這種調控機制和結合方式,可以在體外分別合成此ncRNA和DNA片段,并用同位素(P32)標記,在體外做結合實驗,如果不結合,那么就會呈現兩條已知的條帶,如果能夠結合,那么就會有一條RNA-DNA分子量新條帶出現。
(二)RNA維度
1)作用的ncRNA類型:mRNA-miRNA-lncRNA+circRNA等
2)研究方法
既然是RNA-RNA相互作用,除了胞內FISH定位技術以外,還有其他技術要使用,如下
1.CLASH (crosslinking-ligation and sequencing ofhybrids)
對RNA 分子與RNA 分子相互作用進行研究的實驗系統,主要被用于鑒定miRNA 與其靶mRNA 的相互作用。大概流程為:首先對細胞進行交聯處理,將miRNA 及其靶mRNA 與RISC 復合物固定連接;隨后通過Co-IP富集純化RISC 復合物;之后再通過分子內連接將miRNA 的3'-OH 末端與靶mRNA 的5'-PO4 末端連接,形成一條長的單鏈RNA,然后測序。

2.CHART(CaptureHybridization Analysis of RNA Targets)
也可以叫做RNA precipitation,根據ncRNA序列設計探針,二次探針交聯在固相載體上,若將核內RNA-DNA狀態固定處理,染色質超聲打碎成小片段,RNA-DNA的結構就可以被探針抓取下來,利用DNA-Seq對抓取的DNA進行測序,獲得DNA結合序列的信息(不過這個可以被生信分析工具所預測一下)。
3.熒光素酶報告系統
在熒光素酶報告基因載體的報告基因編碼區的下游插入ncRNA結合序列或者mRNA的3’ UTR區,然后將構建好的載體轉染細胞并改變細胞中相應miRNA的表達水平,通過檢測熒光素酶的表達情況以分析ncRNA或者mRNA的3’ UTR區中是否含有miRNA的靶位點。同理任意兩種RNA-RNA互作都可以利用這個原理。

(三)蛋白質維度
1)作用的ncRNA類型:lncRNA、mRNA較多
2)研究方法:
這種互作模式是RNA-Protein,那么ncRNA可以和調控DNA的蛋白結合,也可以和細胞質內的蛋白質結合,發揮不同效應。
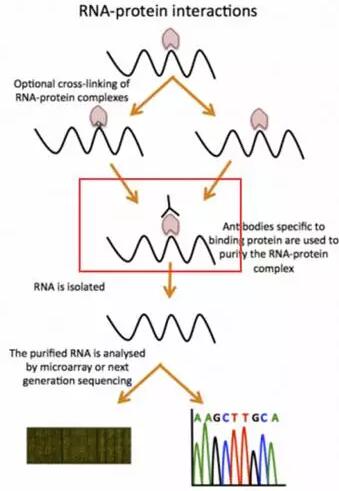
1. CLIP-seq:鑒定與特定RNA結合蛋白相互作用的RNA 分子
CLIP-seq也被稱為HITS-CLIP,是將紫外交聯免疫共沉淀技術(CLIP)與高通量測序技術相結合的技術。
大概流程為:首先通過紫外照射將細胞內的RNA分子與RNA結合蛋白進行耦聯;細胞裂解,免疫共沉淀,核酸酶(RNase)將共沉淀的RNA切割,只保留蛋白質內部的RNA片段;通 過5'末端放射性標記RNA分子及SDS-PAGE電泳,選擇性地回收目的蛋白結合的RNA片段;最后高通量測序。
其可以衍生了許多變異的CLIP-seq方法,例如增強了交聯強度的PAR-CLIP和能夠精確鑒定RNA分子中蛋白質結合位點的iCLIP等。
2.mRNA-蛋白研究方法:利用Poly(A)尾
首先紫外交聯(UV-CL),將mRNA 與蛋白質的相互作用固定連接;隨后使用耦聯磁珠的Oligo(dT) 富集純化mRNA 蛋白復合物,經過洗滌及降解RNA 等步驟,最后將獲得的蛋白質進行質譜分析。
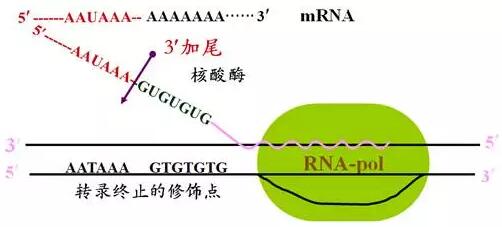
3.EMSA
凝膠遷移或電泳遷移率實驗(EMSA-electrophoretic mobility shift assay)用于研究RNA結合蛋白和特定的RNA序列的相互作用。
通常將純化的蛋白和細胞粗提液和32P同位素標記的RNA探針一同保溫,在非變性的聚丙烯凝膠電泳上,分離復合物和非結合的探針。RNA-復合物比非結合的探針移動得慢。
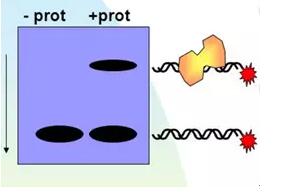
4.RIP(RNA-immuno precipitation)
RNA免疫沉淀可以用來研究組織或活細胞中蛋白質和RNA之間的相互作用。該方法的原理是采用針對目標蛋白的抗體, 把相應的RNA-蛋白復合物沉淀下來, 經過分離純化, 并對復合物上的RNA進行分析鑒定。
RIP技術分別與Chip和RNA-Seq相結合而衍生的RIP-Chip和RIP-Seq技術, 也廣泛應用于lncRNA與蛋白質的相互作用中。
非編碼RNA可火了,miRNA大火燒了十幾年,現在慢慢地把lncRNA的領域也給點著了。現在可能連實驗室的插槍頭阿姨都知道絕大多數的RNA分子并不翻譯,這非編碼RNA(noncoding RNA),包括miRNA、lncRNA等。
目前主要研究方向集中在非編碼RNA 及其相互結合的DNA、RNA、蛋白質等科學問題,而科學問題的解答依賴于實驗技術的創新使用和革命。
IncRNA可以海陸空全面調控細胞功能,厲害了,其實主要指lncRNA,而miRNA、siRNA主要作用于mRNA層面,本文按非編碼RNA的三個調控維度,就當前主要的實驗技術簡單介紹一下。
(一)DNA維度
1)作用的ncRNA類型:lncRNA等
2)研究方法介紹有:
和DNA序列結合的調控目的無非是①阻止下游基因的轉錄②把轉錄因子、甲基化酶等招惹過來修飾DNA序列。他們的共同特點是和解鏈的DNA的某一條鏈結合,那么研究方法包括

1. RNA原位雜交(RNAin situ hybridization)
檢測該ncRNA是否位于核內,如果不在核內,那就不用考慮和DNA作用了。
RNA原位雜交是指將特定標記的已知順序核酸為探針與細胞或組織切片中核酸進行雜交,從而對特定核酸順序進行精確定量定位的過程。原位雜交可以在細胞標本或組織標本上進行。根據RNA序列設計cDNA、cRNA探針,對目標進行定位。

2. ChIRP(chromatin isolation by RNA purifications)
是一項通過純化RNA 分子從而獲得其結合的染色質片段( 包括RNA 結合蛋白與基因組DNA) 的實驗方法。
大概流程:首先細胞交聯,使lncRNA、染色質及被固定連接;隨后通過超聲將染色質打斷,并使用帶有生物素標記的tiling oligos與長鏈非編碼RNA 分子雜交;之后利用親和素磁珠純化長鏈非編碼RNA 及其結合的染色質片段;后續分析和測序。

3. ncRNA-DNA三聯體的體外同位素檢測
為了進一步檢驗這種調控機制和結合方式,可以在體外分別合成此ncRNA和DNA片段,并用同位素(P32)標記,在體外做結合實驗,如果不結合,那么就會呈現兩條已知的條帶,如果能夠結合,那么就會有一條RNA-DNA分子量新條帶出現。

(二)RNA維度
1)作用的ncRNA類型:mRNA-miRNA-lncRNA+circRNA等
2)研究方法
既然是RNA-RNA相互作用,除了胞內FISH定位技術以外,還有其他技術要使用,如下
1.CLASH (crosslinking-ligation and sequencing ofhybrids)
對RNA 分子與RNA 分子相互作用進行研究的實驗系統,主要被用于鑒定miRNA 與其靶mRNA 的相互作用。大概流程為:首先對細胞進行交聯處理,將miRNA 及其靶mRNA 與RISC 復合物固定連接;隨后通過Co-IP富集純化RISC 復合物;之后再通過分子內連接將miRNA 的3'-OH 末端與靶mRNA 的5'-PO4 末端連接,形成一條長的單鏈RNA,然后測序。

2.CHART(CaptureHybridization Analysis of RNA Targets)
也可以叫做RNA precipitation,根據ncRNA序列設計探針,二次探針交聯在固相載體上,若將核內RNA-DNA狀態固定處理,染色質超聲打碎成小片段,RNA-DNA的結構就可以被探針抓取下來,利用DNA-Seq對抓取的DNA進行測序,獲得DNA結合序列的信息(不過這個可以被生信分析工具所預測一下)。

3.熒光素酶報告系統
在熒光素酶報告基因載體的報告基因編碼區的下游插入ncRNA結合序列或者mRNA的3’ UTR區,然后將構建好的載體轉染細胞并改變細胞中相應miRNA的表達水平,通過檢測熒光素酶的表達情況以分析ncRNA或者mRNA的3’ UTR區中是否含有miRNA的靶位點。同理任意兩種RNA-RNA互作都可以利用這個原理。

(三)蛋白質維度
1)作用的ncRNA類型:lncRNA、mRNA較多
2)研究方法:
這種互作模式是RNA-Protein,那么ncRNA可以和調控DNA的蛋白結合,也可以和細胞質內的蛋白質結合,發揮不同效應。
1. CLIP-seq:鑒定與特定RNA結合蛋白相互作用的RNA 分子
CLIP-seq也被稱為HITS-CLIP,是將紫外交聯免疫共沉淀技術(CLIP)與高通量測序技術相結合的技術。
大概流程為:首先通過紫外照射將細胞內的RNA分子與RNA結合蛋白進行耦聯;細胞裂解,免疫共沉淀,核酸酶(RNase)將共沉淀的RNA切割,只保留蛋白質內部的RNA片段;通 過5'末端放射性標記RNA分子及SDS-PAGE電泳,選擇性地回收目的蛋白結合的RNA片段;最后高通量測序。
其可以衍生了許多變異的CLIP-seq方法,例如增強了交聯強度的PAR-CLIP和能夠精確鑒定RNA分子中蛋白質結合位點的iCLIP等。

2.mRNA-蛋白研究方法:利用Poly(A)尾
首先紫外交聯(UV-CL),將mRNA 與蛋白質的相互作用固定連接;隨后使用耦聯磁珠的Oligo(dT) 富集純化mRNA 蛋白復合物,經過洗滌及降解RNA 等步驟,最后將獲得的蛋白質進行質譜分析。
3.EMSA
凝膠遷移或電泳遷移率實驗(EMSA-electrophoretic mobility shift assay)用于研究RNA結合蛋白和特定的RNA序列的相互作用。
通常將純化的蛋白和細胞粗提液和32P同位素標記的RNA探針一同保溫,在非變性的聚丙烯凝膠電泳上,分離復合物和非結合的探針。RNA-復合物比非結合的探針移動得慢。

4.RIP(RNA-immuno precipitation)
RNA免疫沉淀可以用來研究組織或活細胞中蛋白質和RNA之間的相互作用。該方法的原理是采用針對目標蛋白的抗體, 把相應的RNA-蛋白復合物沉淀下來, 經過分離純化, 并對復合物上的RNA進行分析鑒定。
RIP技術分別與Chip和RNA-Seq相結合而衍生的RIP-Chip和RIP-Seq技術, 也廣泛應用于lncRNA與蛋白質的相互作用中。

最新動態
-
09.23
中藥的現代詮釋:外泌體如何革新傳統醫學?
-
07.02
1+1>2!深度解析RNA測序數據挖掘邏輯和后期實驗設計思路,輕松研獲10+ SCI
-
07.01
“稻”亦有道——盤點近期水稻研究的重大突破
-
06.28
科學與美學的結合體:植物亞細胞定位技術詳解
-
06.28
“聚焦新質生產力,激發科研新動能”|LCA躋身蛋白互作研究的新銳力量
-
06.05
知無不“研”|一文讀懂免疫共沉淀技術(Co-IP)
-
05.14
四大研究利器(Co-IP、BIFC、Y2H、GST pull-down)助力速配蛋白互作“最佳拍檔”
-
05.14
高效、精準、直觀、實時——取經“蛋白互作研究翹楚”BIFC!
-
05.14
轉染效率低、干擾效果差、重復性欠佳...siRNA研究頻遇“攔路虎”怎么辦?
-
04.22
一文讀懂EMSA技術核心要點,讓“emsa” 秒變“easy”










