解析腫瘤Hi-C多組學研究策略,IF≥10+文章不是夢!
信息來源:金開瑞 作者:genecreate 發布時間:2018-06-29 09:46:43
Hi-C (High-through chromosome conformation capture) 是以整個細胞核為研究對象,利用高通量測序技術,結合生物信息分析方法,研究全基因組范圍內整個染色質DNA在空間位置上的關系,獲得高分辨率的染色質調控元件相互作用圖譜。Hi-C可以與RNA-Seq、ChIP-Seq、ATAC-Seq等數據進行聯合分析,從基因調控網絡和表觀遺傳網絡來闡述生物體性狀形成的相關機制。
今天,小編以一篇經典文獻帶您了解Hi-C技術在疾病發生機制中的應用。
題目:Chromatin interaction analysis reveals changes in small chromosome and telomere clustering between epithelial and breast cancer cells
期刊:Genome Biology
影響因子:11.908
研究目的:采用HI-C技術探究乳腺癌細胞三維空間結構改變及其致病機制
實驗分組:人乳腺上皮細胞系MCF-10A和乳腺癌細胞系MCF-7
測序策略:Hi-C:HiSeq 2000 PE100;RNA-seq:HiSeq 2000 SE100
研究思路:
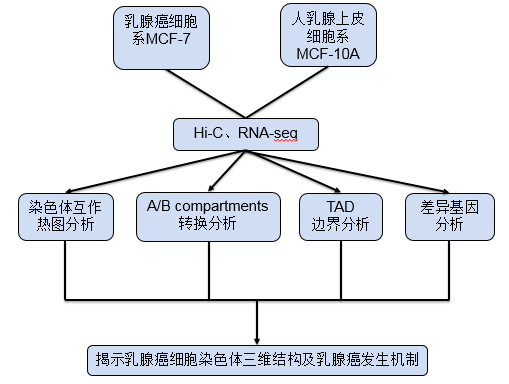
研究背景
三維基因組對于通過將遠距離啟動子、增強子和其他順式調控區域集中在一起,來調控基因表達是非常重要的。癌癥的發展導致異常基因表達的幾種遺傳和表觀遺傳改變。 此外,癌癥是一種疾病,其細胞核中的主要形態學發生變化,用作診斷標記。盡管癌癥的形態學特征具有很好的特征,但異常核形態的分子結構仍然不甚了解。
核內染色質的高級折疊涉及跨越不同尺度的分層結構。顯微成像顯示染色體位于被稱為染色體區域的限定區域內。在細胞核中,每條染色體都有一個優先但不固定的位置,其中基因密集的染色體往往位于核內部。越來越多的證據強調了乳腺癌啟動過程中染色體和基因定位的重要性。此外,最近的數據表明核的物理空間接近度對復發性易位的影響。
幾項研究表明,基因組劃分為兩個區域,稱為A/B compartments,它們在基因組中一般呈間隔分布。研究發現A compartment通常與表達活躍的open chromatin相關,而B compartment與轉錄抑制的closed chromatin相關。
TAD(topologically associating domains)是拓撲關聯域,一般指具有“特殊生物學功能”的相互作用“方塊”,一般區域內部的互作頻率顯著高于毗鄰的兩個區域的互作頻率,是基因組在空間結構中基本的組織形式。已顯示TAD在不同物種,細胞類型和生理條件下基本不變,并可作為轉錄調控的功能單位。最近的研究闡明了TAD和轉錄因子相關的相互作用在激素調節(即雌激素或黃體酮治療)背景下基因組范圍內的作用。TAD被認為通過整合相同領域的監管活動促進轉錄調控。在TAD中,10 kb至1 Mb范圍內的循環相互作用將增強子和啟動子組合在一起以調節基因表達。盡管之前的研究數量眾多,但正常上皮細胞和致瘤性乳腺癌細胞之間全基因組染色質結構的差異仍然未知。
在本研究中,為了表征乳腺癌發展過程中基因組的不同結構,作者在MCF-10A乳腺上皮細胞系和MCF-7乳腺癌細胞系中進行了全基因組染色質構象捕獲(Hi-C)分析。 Hi-C是探測全基因組染色質相互作用的強大分子工具。研究結果揭示了兩種常用的乳腺上皮細胞和致瘤性乳腺癌細胞系在不同分辨率上的染色質結構的差異。這項工作為了解乳腺癌染色質結構改變與基因表達之間的關系奠定了重要的基礎。
研究內容及結果
1. 染色質相互作用熱圖分析比較
為了探測乳腺上皮細胞和乳腺癌細胞的全基因組染色質結構,作者從MCF-10A和MCF-7細胞系分別構建了來自兩個獨立生物學重復樣本的Hi-C文庫,在生物學重復之間分析結果具有高度的一致性。結果發現人乳腺上皮細胞系MCF-10A,在chr16-chr22號小染色體中,有較強的相互作用。
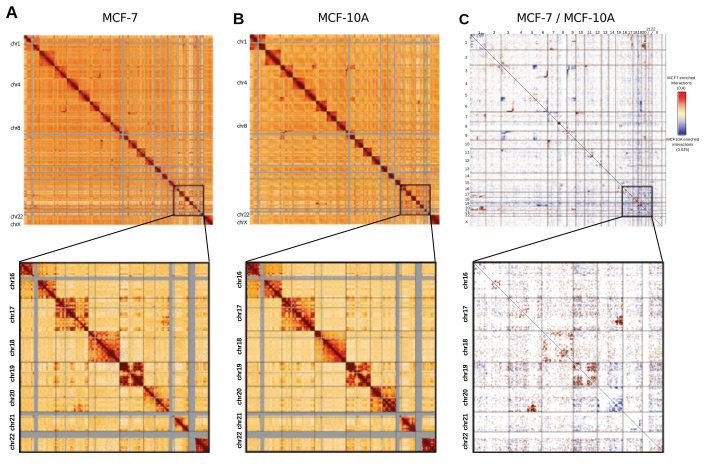
MCF-10A和MCF-7中chr16-chr22號染色體間互作比較
2. A/B compartment互相轉化分析
基因組中存在兩種獨特的相互作用模式,代表開放(A型)和封閉(B型)基因組區室。作者確定了兩種基因組中的區域化模式在生物學重復中具有高重現性。在250 kb的分辨率下,MCF-10A和MCF-7基因組顯示出開放和閉合區室的相似分布。只有一些區域有A到B區室或者B到A區室的轉化,這些轉化區域中的很多基因與癌癥重要通路WNT相關。
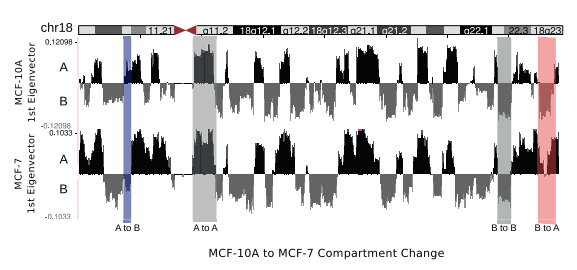
A/B compartment 互相轉化分析
3. TAD邊界分析
盡管染色體結構以及區域化和基因表達都有所變化,但不同細胞系中約有85%(2805)的TAD邊界是相同的。這一結果表明,盡管具有細胞類型特異性和大規模結構差異,但TAD邊界在非致瘤細胞和致瘤細胞之間是一致的。同時,用40kb分辨率來鑒定TAD邊界,發現一些TAD邊界是乳腺癌細胞系特有的。
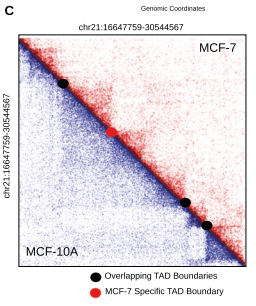
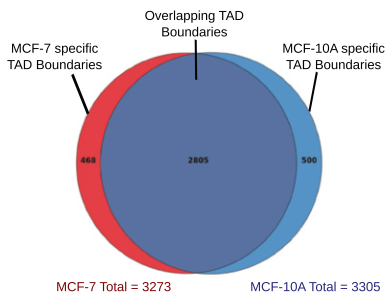
TAD邊界分析
總體而言,在本研究中,從大規模染色體順式和反式相互作用到基因組區域化和TAD形成,作者繪制了乳腺上皮細胞和乳腺癌細胞在不同分辨率下的染色質結構,將會對染色質結構在轉錄調控和腫瘤發生中的功能作用提供更多的依據。
文章小結:作者展示了乳腺上皮細胞和乳腺癌細胞之間染色體結構和基因表達之間的復雜關系。更重要的是,這項研究繪制了染色質高級結構的全基因組視圖,并提供了一種渠道,該渠道可以用于研究乳腺癌的兩種細胞系中的染色質相互作用。
解析文獻
A. Rasim Barutcu1, Bryan R. Lajoie2, et al. Chromatin interaction analysis reveals changes in small chromosome and telomere clustering between epithelial and breast cancer cells. Genome Biology, 2015, 16:214.
參考文獻
1. Bolzer A, Kreth G, et al.Three-dimensional maps of all chromosomes in human male fibroblast nuclei and prometaphase rosettes. PLoS Biol. 2005;3:e157.
2. Imakaev M, Fudenberg G, et al. Iterative correction of Hi-C data reveals hallmarks of chromosome organization. Nat Methods. 2012;9:999–1003.
3.Bickmore WA. The spatial organization of the human genome. Annu Rev Genomics Hum Genet. 2013;14:67–84.
4. Rousseau M, Ferraiuolo MA, et al. Classifying leukemia types with chromatin conformation data. Genome Biol. 2014;15:R60.
5. Dixon JR, Jung I, et al. Chromatin architecture reorganization during stem cell differentiation. Nature. 2015;518:331–6.
最新動態
-
09.23
中藥的現代詮釋:外泌體如何革新傳統醫學?
-
07.02
1+1>2!深度解析RNA測序數據挖掘邏輯和后期實驗設計思路,輕松研獲10+ SCI
-
07.01
“稻”亦有道——盤點近期水稻研究的重大突破
-
06.28
科學與美學的結合體:植物亞細胞定位技術詳解
-
06.28
“聚焦新質生產力,激發科研新動能”|LCA躋身蛋白互作研究的新銳力量
-
06.05
知無不“研”|一文讀懂免疫共沉淀技術(Co-IP)
-
05.14
四大研究利器(Co-IP、BIFC、Y2H、GST pull-down)助力速配蛋白互作“最佳拍檔”
-
05.14
高效、精準、直觀、實時——取經“蛋白互作研究翹楚”BIFC!
-
05.14
轉染效率低、干擾效果差、重復性欠佳...siRNA研究頻遇“攔路虎”怎么辦?
-
04.22
一文讀懂EMSA技術核心要點,讓“emsa” 秒變“easy”










