利用iTRAQ定量蛋白質組學篩選與DAI相關的生物標志物
信息來源:金開瑞 作者:genecreate 發布時間:2018-09-05 16:53:55
Itraq定量蛋白質組學技術和生物信息學分析鑒定大鼠彌漫性軸索損傷的血漿標志物
題目:Identification of plasma biomarkers for diffuse axonal injury in rats by iTRAQ-coupled LC–MS/MS and bioinformatics analysis
期刊:Brain Research Bulletin
影響因子:3.440
合作技術:iTRAQ
研究背景
DAI(彌漫性軸索損傷)是一種嚴重且復雜的腦損傷,目前臨床上沒有相關可靠的生物標志物幫助其進行早期DAI診斷。因此,作者建立了DAI大鼠模型,并利用iTRAQ定量蛋白質組學技術篩選與DAI相關的血漿生物標志物,該研究既有助于DAI臨床診斷的發展,也對DAI潛在分子機制有了進一步了解。
研究內容結果
1. 大鼠腦組織病理學檢查
作者采用HE染色法和Bielschowsky神經纖維銀染法對構建的DAI大鼠模型(1h、6h、1d、3d、7d)進行腦組織病理學檢查 ,結果顯示對照組腦組織樣本未發生任何類型的病理改變,模型組的胼胝體中則觀察到不同程度的軸突腫脹、紊亂、斷開及軸縮球(圖1)。這些結果證實,研究中使用的大鼠樣本適合于進行iTRAQ的定量蛋白質組學分析以鑒定大鼠中的DAI生物標志物。
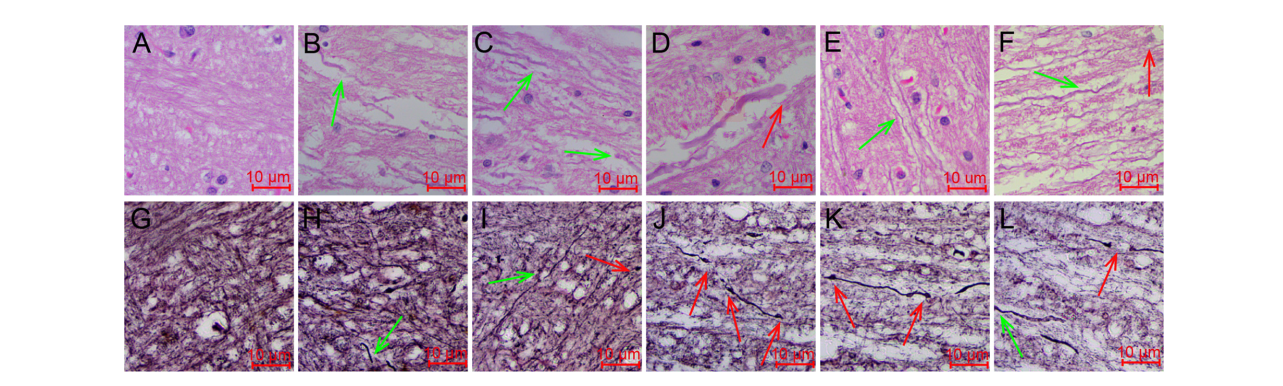
圖1 大腦組織樣本HE染色(A-F)和Bielschowsky神經纖維銀染色(G-L)結果
2. DAI大鼠模型蛋白質組學結果
在對血漿樣品進行iTRAQ蛋白質組學分析中,共鑒定到374個蛋白質,其中共有58個顯著差異蛋白(FC > 1.5,p-values<0.05),和對照組相比,不同時期的模型組中差異蛋白數目如圖2。針對這些差異蛋白,作者進行了GO功能分析和KEGG Pathway分析(圖3)。根據58個差異蛋白的層次聚類分析結果,在3d和7d中的蛋白質豐度發生顯著改變(圖4A)。這可能是因為DAI的發病機制復雜,涉及多階段的生物學過程,軸突損傷1d后可能與不同的分子機制有關。為了研究不同差異蛋白的相互作用,作者又進行了蛋白互作網絡分析,結果顯示GAPDH和Hpx等幾種蛋白質位于蛋白質功能相互作用網絡的中心位置(圖4B)。
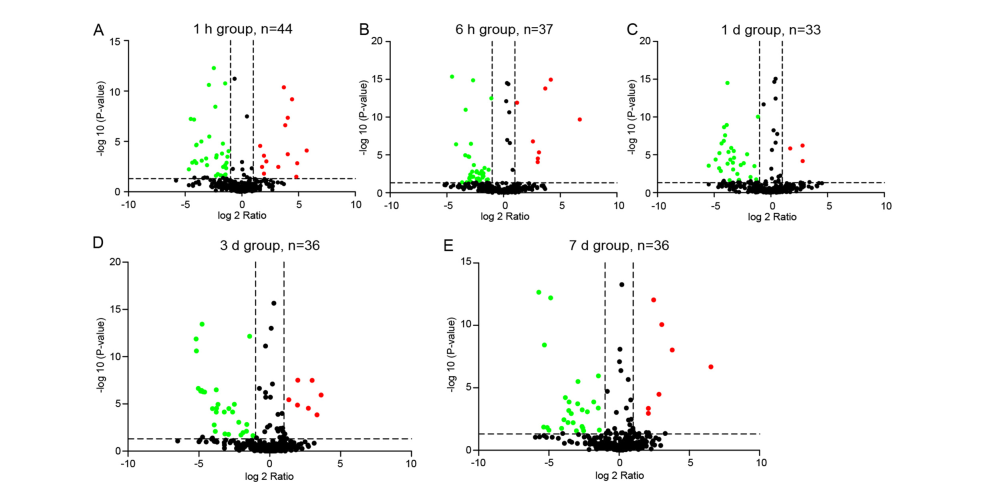
圖2 不同DAI時期蛋白表達火山圖
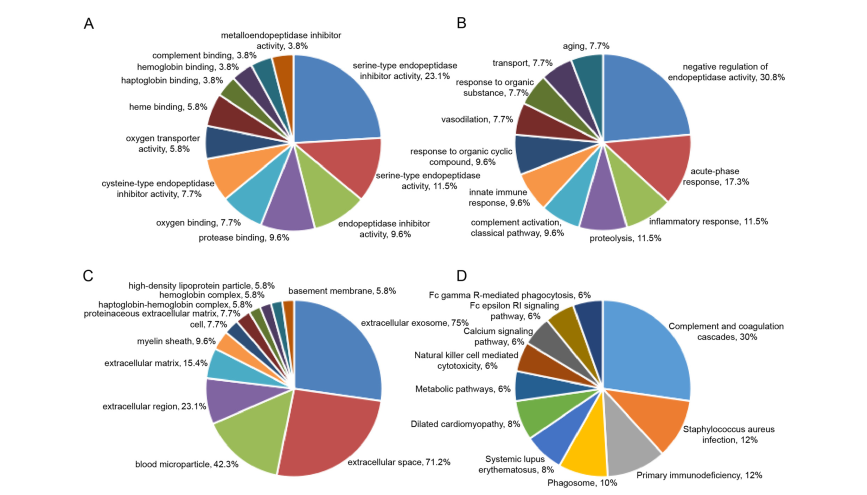
圖3 差異蛋白GO功能分析和KEGG Pathway 分析
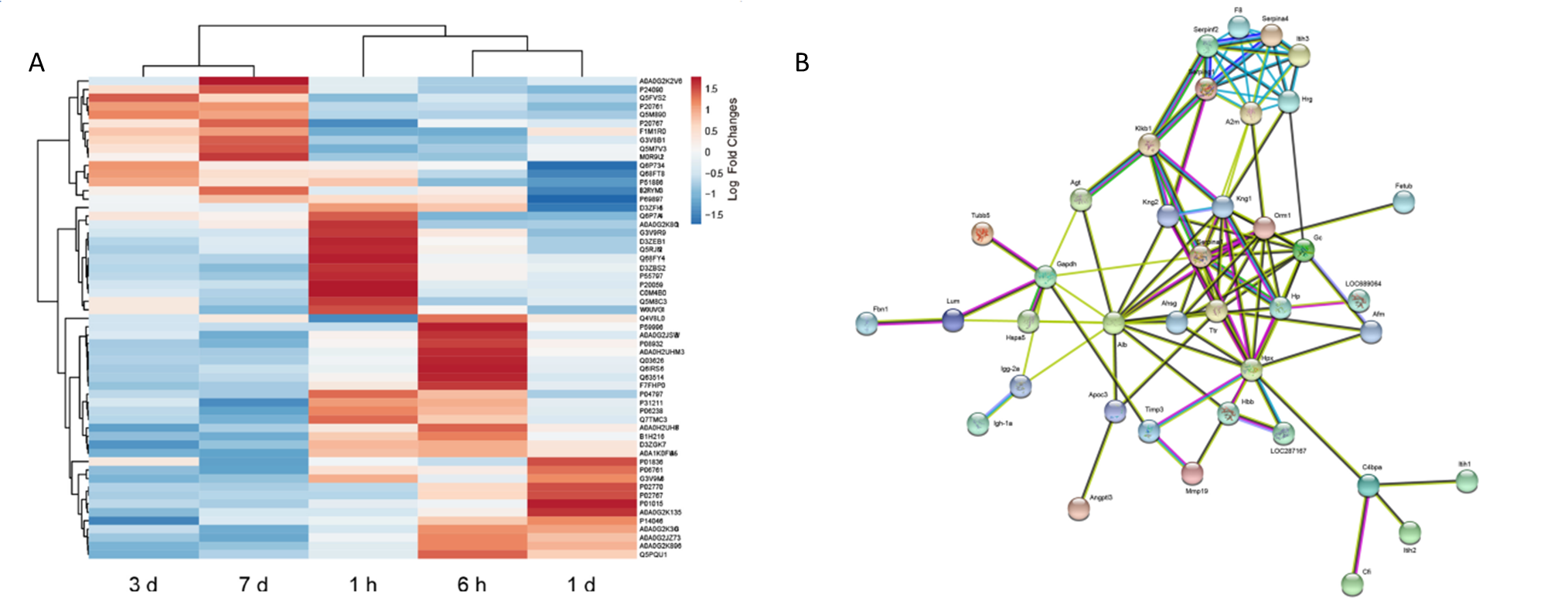
圖4 差異蛋白層次聚類分析和蛋白互作網絡分析
3. 候選標志物驗證
基于蛋白質組學結果,結合蛋白在DAI期間軸突損傷過程中的表達量發生極大變化和潛在的生物學功能,作者選擇GAPDH和Hpx作為候選標志物。和對照組相比,Hpx的表達量在損傷后6h、1d、3d、7d中都顯著下調;GAPDH表達量在損傷后1h、6h顯著上調,隨后又在1d、3d、7d中顯著下降。WB結果與蛋白質組學結果一致(圖5)。
Hpx是一種豐富的血漿糖蛋白,對血紅素有極高的親和力,已有研究證明敲除Hpx會加重無基質血紅蛋白誘導的腦損傷,動物實驗和人神經母細胞瘤細胞培養實驗均顯示Hpx提供針對血紅素活性氧(ROS)毒性的神經保護作用。腦損傷后Hpx表達降低可能加重DAI患者的腦損傷,最終導致死亡。在本研究中,DAI模型的Hpx表達水平在6h、1d、3d和7d組中顯著低于對照組,而在1h組中沒有顯著變化,這意味著Hpx可能參與了非斷離性軸索損傷的生物學過程。這些發現還表明,在DAI中促進Hpx的表達可能有助于減少軸突斷裂和提高臨床療效。
GAPDH是參與糖酵解過程的重要酶,已有研究報道TBI(創傷性腦損傷)后GAPDH顯著降低,同時也會導致能量需求增加。腦損傷后GAPDH顯著下調可導致能源供應的早期減少,最終導致能源供需之間的不平衡。但是下調的GAPDH是否參與非斷離性軸索損傷中的分子和細胞級聯仍有待確定。這些研究結果表明,適當改善能量供應可能有助于減少軸索損傷并獲得更好的DAI患者預后效果。
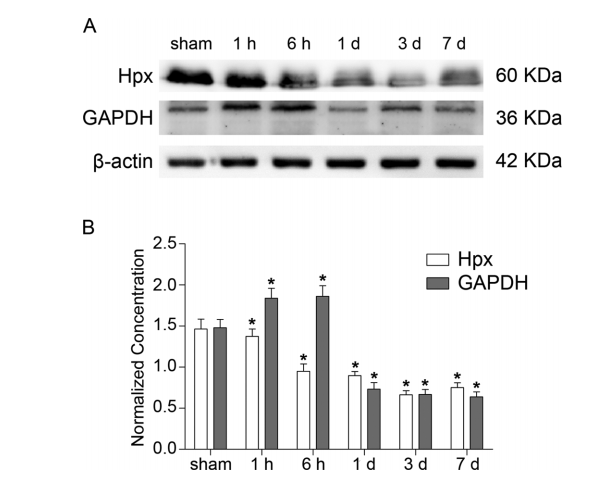
圖5 western blot 驗證候選標志物
研究結論
在本研究中,作者構建了DAI大鼠模型,并應用iTRAQ定量蛋白質組學方法篩選與DAI相關的差異表達的血漿蛋白。基于蛋白質組學結果和蛋白的生物學功能,作者篩選得到了兩個蛋白(Hpx和GAPDH)作為潛在的診斷生物標志物,它們可能在DAI的軸突損傷的生物過程中發揮重要作用。作者的研究結果不僅為DAI的分子機制提供了新的見解,而且還促進了DAI臨床診斷生物標志物組的開發。
解析文獻
Peng Zhanga, Shisheng Zhu, et al. Identification of plasma biomarkers for diffuse axonal injury in rats by iTRAQ-coupled LC–MS/MS and bioinformatics analysis. Brain Research Bulletin, 2018 , 140.
參考文獻
1. Opii, W.O., Nukala, V.N., et al. Proteomic identification of oxidized mitochondrial proteins following experimental traumatic brain injury. J. Neurotrauma.2007, 24 (5), 772–789.
2. Ren, J., Zhao, G., et al. Identification of plasma biomarkers for distinguishing bipolar depressionfrom major depressive disorder by iTRAQ-coupled LC–MS/MS and bioinformatics analysis.Psychoneuroendocrinology .2017, 86, 17–24.
3. Cheow, E.S.H., Cheng, W.C., et al. Myocardial injury is distinguished from stable angina by a set of candidate plasma biomarkers identified using iTRAQ/MRM-based approach. J. Proteome Res. 2018, 17 (1), 499–515.
4. Zhang, P., Zhang, L., et al. Quantitative proteomic analysis to identify differentially expressed proteins in the myocardium of epilepsy using iTRAQ coupled with nano-LC–MS/MS. J. Proteome Res. 2018, 17 (1), 305–314.
最新動態
-
09.23
中藥的現代詮釋:外泌體如何革新傳統醫學?
-
07.02
1+1>2!深度解析RNA測序數據挖掘邏輯和后期實驗設計思路,輕松研獲10+ SCI
-
07.01
“稻”亦有道——盤點近期水稻研究的重大突破
-
06.28
科學與美學的結合體:植物亞細胞定位技術詳解
-
06.28
“聚焦新質生產力,激發科研新動能”|LCA躋身蛋白互作研究的新銳力量
-
06.05
知無不“研”|一文讀懂免疫共沉淀技術(Co-IP)
-
05.14
四大研究利器(Co-IP、BIFC、Y2H、GST pull-down)助力速配蛋白互作“最佳拍檔”
-
05.14
高效、精準、直觀、實時——取經“蛋白互作研究翹楚”BIFC!
-
05.14
轉染效率低、干擾效果差、重復性欠佳...siRNA研究頻遇“攔路虎”怎么辦?
-
04.22
一文讀懂EMSA技術核心要點,讓“emsa” 秒變“easy”










