什么是Hi-C技術(三維基因組)
信息來源:金開瑞 作者:genecreate 發布時間:2022-11-01 15:10:24
染色體由DNA與組蛋白共同組成,從染色體的一級結構(繩珠模型)到四級超螺旋折疊結構,DNA分子一共被壓縮了8400倍左右,正是這些折疊和壓縮,導致基因在細胞中的分布復雜而又有序。
基于基因組序列,調控元件和相關的注釋的信息,科學家們發現,它們在空間結構上并不是在染色體上呈線性地一字依次排開,這些離散的調控元件并不能有效地解釋很多基因的調控結果和機制。由此猜測其與基因組的三維空間結構相關。
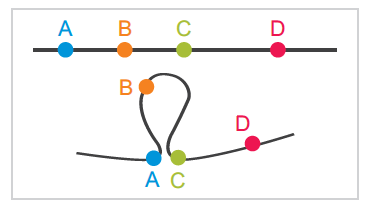
圖1 loops環
基因組三維空間結構與功能的研究簡稱三維基因組學(Three-Dimensional Genomics, 3D Genomics)。2003年Job Dekker及其合作者提出了染色質構象捕獲技術(Chromatin Conformation Capture, 3C),用于測定特定的點到點之間的染色質交互作用。隨后,科學家們擴展了3C技術,開發了4C技術(Circularized Chromatin Conformation Capture),用于測定一點到多點之間的染色質交互作用。Dostie等人接著開發了5C技術(Carbon-Copy Chromatin Conformation Capture),用于測定多點到多點之間的染色質交互作用。
為了能捕獲全基因組范圍的染色質相互作用,Job Dekker研究組又開發出了現在大家所熟知的Hi-C技術。其是基于將線性距離遠、空間結構近的 DNA 片段進行交聯,并將交聯的 DNA 片段富集,接著進行高通量測序,對測序數據分析可以揭示染色質的遠程相互作用,從而推導出基因組的三維空間結構和可能的基因之間的調控關系

圖2 Hi-C 實驗流程示意圖
(引自Lieberman-Aiden E, van Berkum NL, et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 2009, 326(5950):289-93)

圖3 序列裝配過程圖
(引自TingXie, Jue-FeiZheng, et al. De Novo Plant Genome Assembly Based on Chromatin Interactions: A Case Study of Arabidopsis thaliana. Molecular Plant, 2015, 489-492)
我們優化的Hi-C技術
武漢金開瑞生物工程有限公司技術顧問、華中農業大學教授, 曹罡課題組與華中農業大學李國亮教授課題組在國際著名期刊Nature Genetic(IF=27.125)上聯合發表題為“Digestion-ligation-only Hi-C is an efficient and cost-effective method for chromosome conformation capture”的研究論文。該論文主要介紹了一種新的染色體構象捕獲技術(DLO Hi-C),此技術信噪比高,質量控制于早期,為解析基因組三維結構提供了一種新型、高效、經濟的研究方法。

圖4 DLO Hi-C實驗流程
新的DLO Hi-C染色體構象捕獲技術(digestion-ligation-only Hi-C,DLO Hi-C),設計了巧妙的酶切位點,采用同時酶切酶連的方式,將DNA接頭連接在染色體內切酶切口末端上,然后進行鄰近酶連,最后再用MmeI內切酶酶切消化,回收固定大小互作DNA片段。此方法具有以下特點:1. 雙交聯,兩次連接和消化,其中第一次同時酶切酶連,收集測序固定片段;2. 無稀釋液體交聯,無生物素。
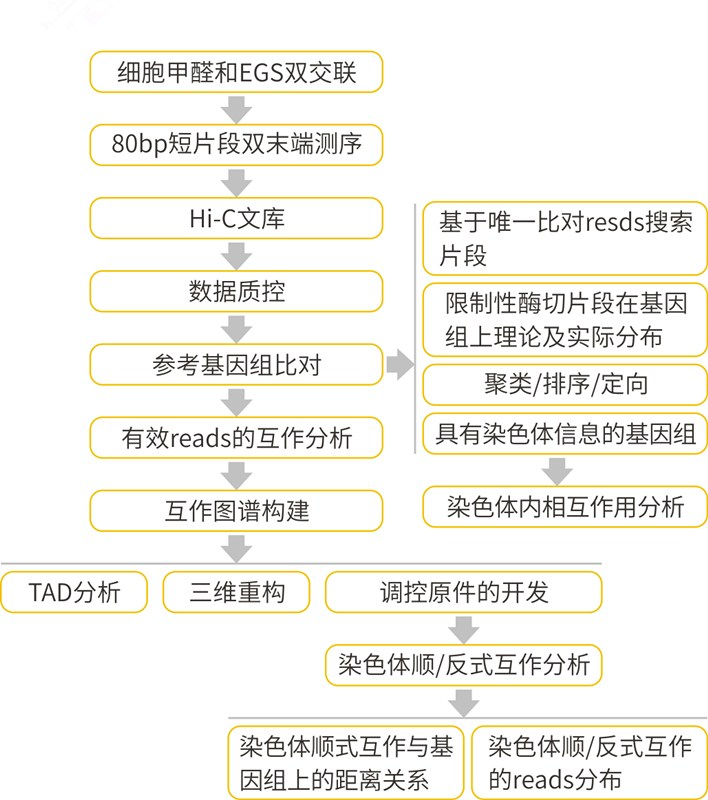
圖5 Hi-C生信分析流程

圖6 Hi-C多組學分析
DLO Hi-C和其他Hi-C的衍生技術的結果比對
如表1所示,DLO Hi-C技術所能獲取的圖譜分辨率高于傳統Hi-C技術、DNase Hi-C技術。在非冗余的有效數據讀取中in situ DLO Hi-C的讀取效率和in situ Hi-C相似。
表1
幾種不同的Hi-C衍生方法的對比分析

(引自Lin D, Hong P, et al. Digestion-ligation-only Hi-C is an efficient andcost-effective method for chromosome conformation capture. Nature Genetics, 2018 , 50 (5))
最終所呈現的圖譜如圖7所示。in situ DLO Hi-C和in situ Hi-C所呈現的矩陣清晰度相似,分辨率較高。

圖7 幾種不同的Hi-C的衍生方法的矩陣圖
(引自Lin D, Hong P, et al. Digestion-ligation-only Hi-C is an efficient andcost-effective method for chromosome conformation capture. Nature Genetics, 2018 , 50 (5))
三種Hi-C 技術在讀取A/B compartments數據時,無論原始數據量多大,所讀取的數據相似,且2787個數據在三種技術中均被一致讀取。

圖8 三種Hi-C技術關于A/B compartments的讀取
(引自Lin D, Hong P, et al. Digestion-ligation-only Hi-C is an efficient andcost-effective method for chromosome conformation capture. Nature Genetics, 2018 , 50 (5))
三種Hi-C 技術在讀取TADs數據時,in situ DLO Hi-C所讀取的TADs區最多是3244個。DLO Hi-C的TADs區的讀取率最高,且三種技術讀取了2334個一致的TADs數據。

圖9 三種Hi-C技術關于TADs的讀取
(引自Lin D, Hong P, et al. Digestion-ligation-only Hi-C is an efficient andcost-effective method for chromosome conformation capture. Nature Genetics, 2018 , 50 (5))
三種Hi-C 技術在讀取Loops數據時,in situ DLO Hi-C所讀取的loops最多是12780個。這三種技術共同讀取的Loops較少,929個。

圖10 三種Hi-C技術關于Loops的讀取
(引自Lin D, Hong P, et al. Digestion-ligation-only Hi-C is an efficient andcost-effective method for chromosome conformation capture. Nature Genetics, 2018 , 50 (5))
綜上所述,DLO Hi-C技術可獲取的有效的制作矩陣的數據、A/B compartments數據、TADs數據和Loops數據較優于其他Hi-C的衍生技術。
如此強大的Hi-C,到底該如何運用于生物科學研究中呢?欲知詳情,且聽下回分解!
相關技術服務
推薦閱讀
最新動態
-
09.23
中藥的現代詮釋:外泌體如何革新傳統醫學?
-
07.02
1+1>2!深度解析RNA測序數據挖掘邏輯和后期實驗設計思路,輕松研獲10+ SCI
-
07.01
“稻”亦有道——盤點近期水稻研究的重大突破
-
06.28
科學與美學的結合體:植物亞細胞定位技術詳解
-
06.28
“聚焦新質生產力,激發科研新動能”|LCA躋身蛋白互作研究的新銳力量
-
06.05
知無不“研”|一文讀懂免疫共沉淀技術(Co-IP)
-
05.14
四大研究利器(Co-IP、BIFC、Y2H、GST pull-down)助力速配蛋白互作“最佳拍檔”
-
05.14
高效、精準、直觀、實時——取經“蛋白互作研究翹楚”BIFC!
-
05.14
轉染效率低、干擾效果差、重復性欠佳...siRNA研究頻遇“攔路虎”怎么辦?
-
04.22
一文讀懂EMSA技術核心要點,讓“emsa” 秒變“easy”










