發現蛋白質組和靶向蛋白質組聯合揭示口腔鱗癌預后診斷標志物
信息來源:金開瑞 作者:genecreate 發布時間:2018-11-28 16:25:36
題目:Combining discovery and targeted proteomics reveals a prognostic signature in oral cancer
發現蛋白質組和靶向蛋白質組聯合揭示口腔鱗癌預后診斷標志物
期刊:Nature Communications
影響因子:12.353
主要技術:Label free
研究背景
口腔鱗癌(ral squamous cell carcinoma,OSCC)是頭頸部惡性腫瘤的最常見類型,表現出高患病率和發病率,OSCC不同組織區域具有特定的組織病理學和分子特征,限制了基于腫瘤-結節-轉移(tumor−node –metastasis,TNM)標準的臨床分期,接受標準治療的患者OSCC復發率在18%至76%之間。因此,評估OSCC患者的預后結果的生物學標志物具有重要的臨床意義。
研究內容及結果
1. OSCC蛋白表達譜
作者選取了20例OSCC患者的石蠟包埋組織,并對組織中的6個區域進行Label free定量蛋白質組學檢測分析(圖1,圖2a-b),共定量到2049個蛋白質,發現在ITF(距腫瘤切片邊緣1mm深度組織)和內部腫瘤(距離上皮腫瘤組織起源1mm組織)的腫瘤島存在32個差異表達蛋白,腫瘤基質中存在101個差異表達蛋白。層次聚類分析不僅顯示了來自ITF和內部腫瘤的樣品之間的蛋白質組多樣性,還顯示了腫瘤島蛋白和腫瘤基質蛋白差異(圖2c-d)。為了研究生物過程是否可以在空間上將腫瘤島與腫瘤基質分開,作者進行了GO富集分析,發現腫瘤島蛋白多參與細胞代謝過程,腫瘤基質蛋白多參與細胞粘附過程等(圖2g)。最后,作者結合生信分析和線性回歸分析,確定了14種與病人特征相關的蛋白(表1)。
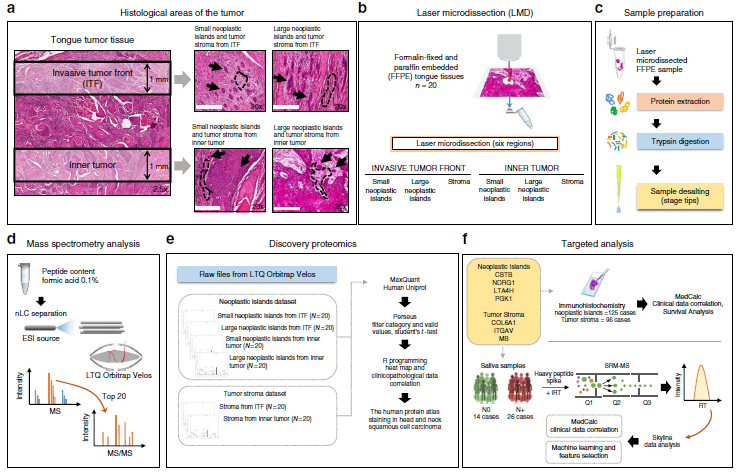
圖1 技術路線
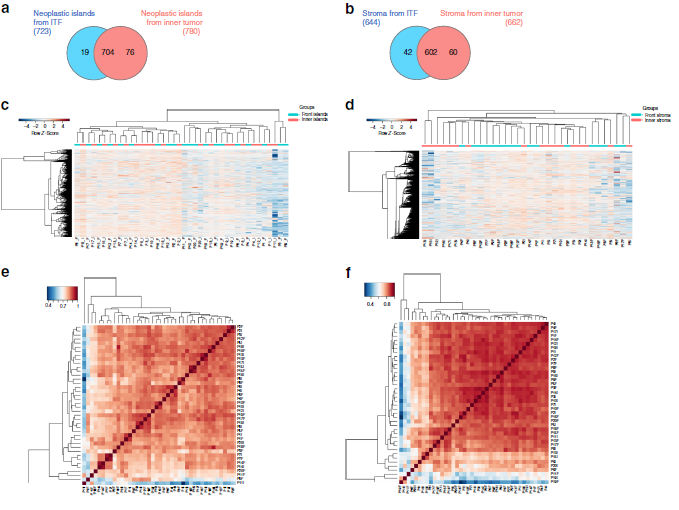
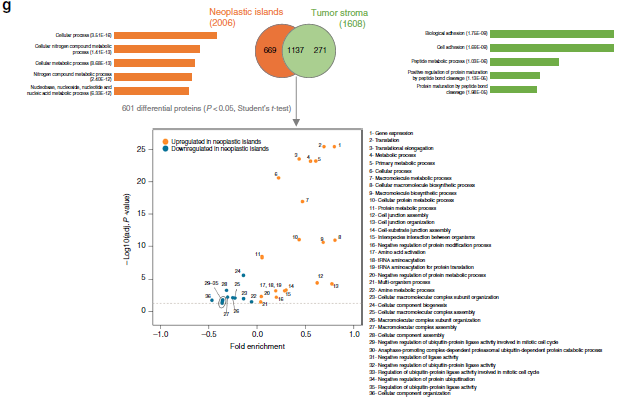
圖2 蛋白質組學分析
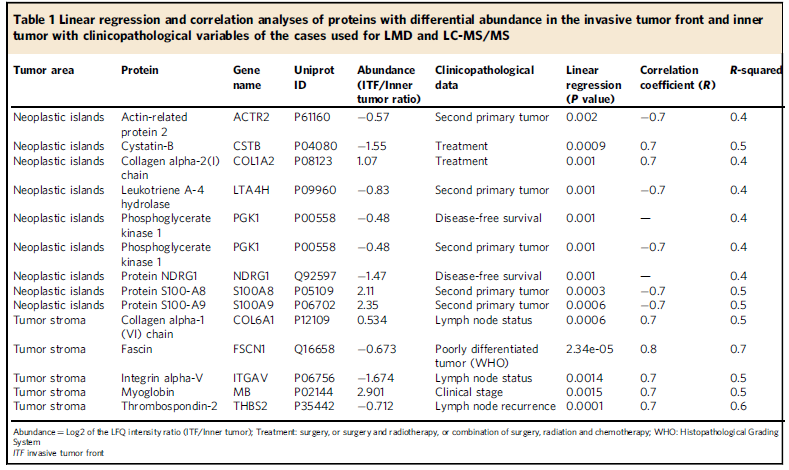
表1 與OSCC相關蛋白列表
2. IHC驗證靶蛋白
上面這些候選蛋白對于后續的IHC和靶向蛋白質組學驗證來說數目仍然較多,因此,作者基于更嚴格的條件進行過濾:(1)在ITF和內部腫瘤存在的差異表達蛋白;(2)與患者臨床特征顯著相關的蛋白質;(3)在人類蛋白質圖譜中具有鱗狀細胞癌陽性染色的蛋白質(https://www.proteinatlas.org/);(4)選擇未見報道或者僅有少數報道跟口腔鱗癌相關的蛋白。基于上述原則,作者共挑選出7個候選蛋白: CSTB、LTA4H、NDRG1、PGK1、COL6A1、ITGAV、MB。
作者首先選擇了原始樣本中的14例OSCC病人石蠟包埋組織對上述7種蛋白進行IHC驗證,結果和蛋白質組學結果一致。隨后,作者將樣本量擴大,包括125例腫瘤島組織和96例腫瘤基質組織進行IHC驗證(圖4)。發現擴大樣本量后大多數IHC結果與原始14例IHC病例相似,但是隨著數據隊列的增加,LTA4H、PGK1和ITGAV染色鑒定區域(ITF或內部腫瘤)略有變化,說明靶蛋白對于其鑒定細胞具有一定的特異性。
為了增加靶蛋白預后診斷標志的可信度,作者將IHC結果與臨床病理學特征結合,發現臨床病理參數和CSTB、PGK1、COL6A1和ITGAV顯著關聯。OSCC患者生存分析表明,ITF中CSTB和NDRG1的低表達分別與局部復發和第二原發腫瘤相關,PGK1和ITGAV的高表達與局部復發和淋巴結復發相關。CSTB、PGK1和ITGAV對于5年疾病特異性存活和無病存活具有顯著性,NDRG1僅對5年無病生存率具有顯著性。
圖3 候選蛋白豐度曲線
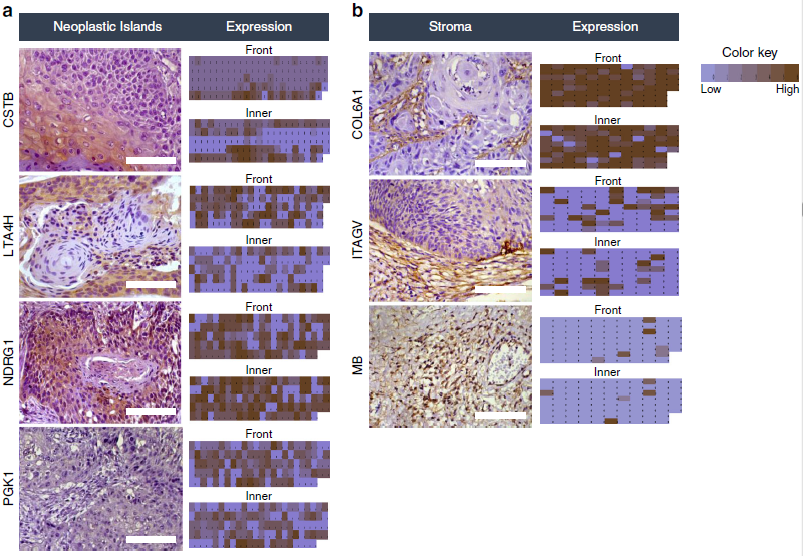
圖4 靶蛋白IHC驗證
3. SRM-MS靶向蛋白質組驗證靶蛋白
接下來作者選取了40例病人唾液對上述7個靶蛋白進行靶向蛋白質組學驗證。樣本共分成了兩組:無淋巴結轉移的患者(N0)和已經發生淋巴結轉移的患者(N+)。結果發現,和N0組相比,有6種蛋白(CSTB、LTA4H、PGKI、NDRG1、COL6A1和ITGAV)在N+中蛋白表達豐度較低(圖5a,c)。此外,作者基于兩種質譜數據采集模式(DDA和SRM)的組織和唾液蛋白之間的關系進行了分析,發現大多數蛋白質在ITF和唾液部位表現出較低的豐度,這與預后不良相關。唾液樣品中LTA4H、PGK1、NDRG1、COL6A1和ITGAV蛋白的低表達與淋巴結轉移和晚期臨床分期相關。
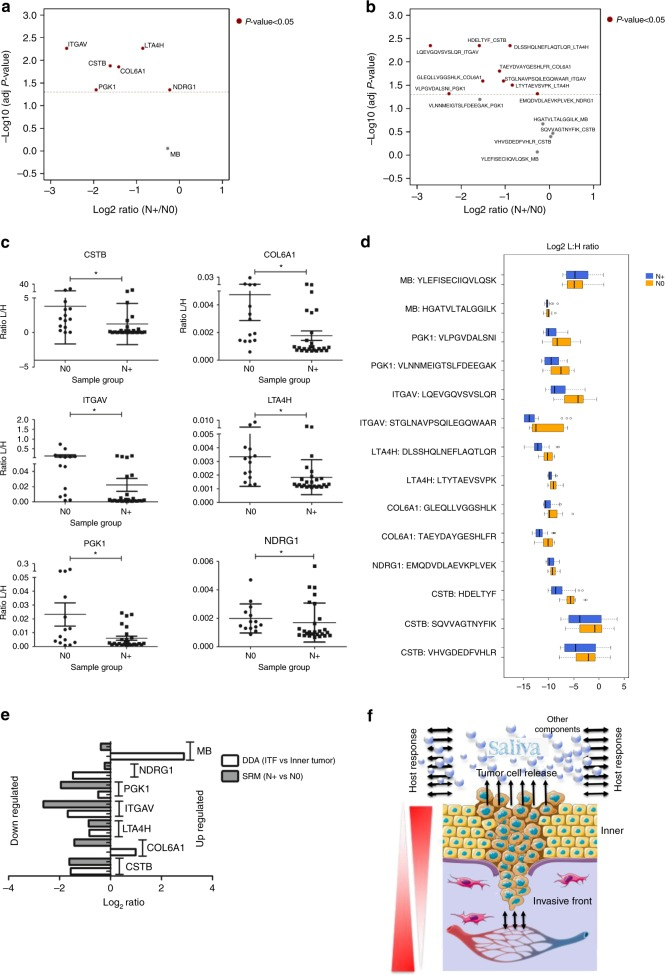
圖5 靶向蛋白質組學驗證
文章小結
發現蛋白質組即定量蛋白質組能夠在空間上繪制腫瘤島及其周圍的OSCC基質的蛋白質組,鑒定具有潛在預后價值的蛋白質,并通過IHC和靶向蛋白質組學SRM-MS進行驗證,有助于推動預后決策,促進精確的治療方案和減少腫瘤局部復發或淋巴結轉移。IHC表明ITF的腫瘤島中CSTB低表達可作為局部復發的獨立標志物。總之,作者運用高通量蛋白質組學分析篩選,靶向蛋白質組學驗證的方法,確定了預后表征蛋白,增強OSCC的預后決策,更好地指導治療,以減少腫瘤復發或淋巴結轉移。
解析文獻
Carolina Moretto Carnielli, Carolina Carneiro Soares Macedo, et al. Combining discovery and targeted proteomics reveals a prognostic signature in oral cancer. Nature Communications, 2018, DOI: 10.1038/s41467-018-05696-2
參考文獻
1. Alfonso, P., Cañamero, M., et al. Proteome analysis of membrane fractions in colorectal carcinomas by using 2D-DIGE saturation labeling. J. Proteome Res, 2008, 7, 4247–4255.
2. Schaaij-Visser, T. B. M. et al. Differential proteomics identifies protein biomarkers that predict local relapse of head and neck squamous cell carcinomas. Clin. Cancer Res, 2009 15, 7666–7675.
3. Chi, L.-M. et al. Enhanced interferon signaling pathway in oral cancer revealed by quantitative proteome analysis of microdissected specimens using 16O/18O labeling and integrated two-dimensional LC-ESI-MALDI tandem MS.Mol. Cell. Proteom, 2009, 8, 1453–1474 .
4. Vizcaíno, J. A. et al. ProteomeXchange provides globally coordinated proteomics data submission and dissemination. Nat. Biotechnol, 2014, 32, 223–226.
5. Winck, F. V. et al. Insights into immune responses in oral cancer through proteomic analysis of saliva and salivary extracellular vesicles. Sci. Rep. 2015, 5, 16305.
6. Harris, T. M. et al. Proteomic analysis of oral cavity squamous cell carcinoma specimens identifies patient outcome-associated proteins. Arch. Pathol. Lab. Med.2015, 139, 494–507.
7. Kawahara, R. et al. A targeted proteomic strategy for the measurement of oral cancer candidate biomarkers in human saliva. Proteomics, 2016, 16, 159–173.
上一條:SWATH-MS的那些事
下一條:簡述外泌體及其蛋白質組學研究
最新動態
-
09.23
中藥的現代詮釋:外泌體如何革新傳統醫學?
-
07.02
1+1>2!深度解析RNA測序數據挖掘邏輯和后期實驗設計思路,輕松研獲10+ SCI
-
07.01
“稻”亦有道——盤點近期水稻研究的重大突破
-
06.28
科學與美學的結合體:植物亞細胞定位技術詳解
-
06.28
“聚焦新質生產力,激發科研新動能”|LCA躋身蛋白互作研究的新銳力量
-
06.05
知無不“研”|一文讀懂免疫共沉淀技術(Co-IP)
-
05.14
四大研究利器(Co-IP、BIFC、Y2H、GST pull-down)助力速配蛋白互作“最佳拍檔”
-
05.14
高效、精準、直觀、實時——取經“蛋白互作研究翹楚”BIFC!
-
05.14
轉染效率低、干擾效果差、重復性欠佳...siRNA研究頻遇“攔路虎”怎么辦?
-
04.22
一文讀懂EMSA技術核心要點,讓“emsa” 秒變“easy”










